dette indlæg blev bidraget af Kurt Thorn fra Nikon Imaging Center på UCSF.
et almindeligt krav til levende celleafbildningseksperimenter er evnen til at følge flere fluorescerende mærkede arter samtidigt. For at gøre det med fluorescerende proteinetiketter kræves flere fluorescerende proteiner, hvis eksitations-og emissionsspektre adskiller sig tilstrækkeligt til, at de kan afbildes i forskellige fluorescerende kanaler på mikroskopet. Med spredning af fluorescerende proteiner i de senere år er der mange fluorescerende proteinkombinationer, der kan afbildes sammen, men det betyder også, at valget af fluorescerende proteiner kræver en vis tanke.

det første skridt i at vælge fluorescerende proteiner til dit multi-color imaging eksperiment er at være opmærksom på, hvad fluorescerende proteiner er tilgængelige. Med nye fluorescerende proteiner, der offentliggøres hver måned, er det en udfordring at beslutte det bedste protein til en given applikation. For at holde dig ajour med de nyeste fluorescerende proteiner opretholder jeg en interaktiv graf og tabel over de bedste fluorescerende proteiner, der i øjeblikket er tilgængelige.
valg af kompatible fluorescerende proteiner
for at vælge et sæt fluorescerende proteiner, der skal afbildes sammen, skal du overveje de samme faktorer som når du vælger et individuelt fluorescerende protein (lysstyrke, fotostabilitet og så videre; se det forrige blogindlæg for mere diskussion af disse faktorer). Derudover skal du også vælge fluorescerende proteiner, der kan skelnes fra hinanden, og som kan afbildes med optikken på det eller de mikroskoper, du har til hensigt at bruge. En nøjagtig bestemmelse af, om to fluorescerende proteiner kan adskilles fra hinanden, kræver kendskab til deres eksitations-og emissionsspektre, men en god tommelfingerregel er, at både spidsbelastningsbølgelængderne og spidsbelastningsbølgelængden for de to proteiner skal adskilles med 50-60 nm. For eksempel kan CFP (eks 430 nm / em 474 nm) og YFP (eks 514 nm / em 527 nm) afbildes sammen, men CFP og GFP (eks 488 nm / em 507 nm) viser noget krydstale mellem de to fluorescerende proteiner. Hvis du skal afbilde fluorescerende proteiner, hvis spektre overlapper hinanden, der er teknikker, som spektral unblanding, som kan bruges til at adskille de fluorescerende proteiner, men disse er uden for rammerne af dette indlæg.
er dine fluorescerende proteiner kompatible med din mikroskopoptik?
for at afgøre, om de fluorescerende proteiner, du er interesseret i, er kompatible med din mikroskopoptik, vil du sammenligne spændings-og emissionsspektre for dit protein med filtersættene eller laserne på dit mikroskop. Ideelt set vil du gerne have en betydelig overlapning mellem eksitations-og emissionsfiltre og eksitations-og emissionsspektre for proteinet, så proteinet er godt ophidset af dit mikroskop, og fluorescensemissionen af proteinet opsamles effektivt af mikroskopet. For at sammenligne kampen mellem et fluorescerende protein og et filtersæt leverer mange filtersætleverandører værktøjer til at plotte fluorescensspektre af proteiner og farvestoffer og deres filtre (se Chroma ‘er, Semrocks eller Omega’ er). Selvom disse ikke indeholder alle fluorescerende proteiner i almindelig brug (især ikke de senest offentliggjorte), kan de være et godt udgangspunkt. I mange tilfælde er det tilstrækkeligt at bruge et spektrum til et nært beslægtet protein, hvis du ved, at dit protein af interesse har et lignende spektrum. For eksempel er her et skærmbillede fra Chroma Spectra-seeren, der sammenligner et standard Cy3-eller Rhodaminfiltersæt (Chroma #49004) til spektre for både mCherry og TagRFP.
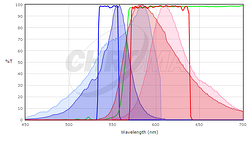
Her vises TagRFP-spektret i de mørkere farver, og mcherry-spektret vises i de lysere farver; eksitationsspektre er blå og emissionsspektre er røde. Hverken er et perfekt match til filtersættet, men eksitationsfilteret ophidser mere af toppen af TagRFP-eksitationen, og emissionsfilteret samler en større brøkdel af TagRFP-emissionen end mCherry-emissionen. For dette filtersæt forventer vi, at TagRFP giver et lysere signal end mCherry. Generelt vil filtersæt designet til Rhodamin / Cy3 fungere bedre med kortere bølgelængde røde fluorescerende proteiner som TagRFP eller mRuby2 end længere bølgelængdeproteiner som mCherry. For baggrund på fluorescens og filter sæt, se Introduktion til fluorescens mikroskopi foredrag på iBiology.
almindeligt anvendte filtersæt & relevante fluorescerende proteiner
almindeligt anvendte filtersæt til flerfarvet billeddannelse inkluderer dem, der er designet til CFP, YFP og RFP eller Sedat-Firfiltersættet, designet til DAPI / Fluorescein / Rhodamin / Cy5 (f. eks. Semrocks) og den lignende 4-laserkombination på en konfokal 405 / 488 / 561 / 640 nm). I vores hænder er de bedste fluorescerende proteiner til billeddannelse med dette sæt mTagBFP2, EGFP eller en af de forbedrede GFP-varianter, mRuby2 eller TagRFP-T og et infrarødt fluorescerende protein som iFP1.4 eller iFP2.0. Pas på, at disse infrarøde fluorescerende proteiner kræver biliverdin som en cofaktor, og så skal du muligvis supplere dine celler med biliverdin for maksimal lysstyrke. I pattedyrceller er en af de forbedrede foldevarianter af EGFP som mEmerald eller Clover sandsynligvis bedst; mNeonGreen er et endnu nyere grønt fluorescerende protein, der formodes at være ekstremt lyst. I S. cerevisiae, vi har testet en række grønne og røde fluorescerende proteiner med dette filtersæt og har rapporteret lysstyrkemålinger. Her overgår EGFP de forbedrede foldevarianter, formodentlig på grund af den lavere væksttemperatur. Dette antyder dog også, at der ikke er noget enkelt fluorescerende protein, der er optimalt for alle organismer, og at hvis du vil have det lyseste signal, skal du muligvis prøve flere proteiner i dit interessesystem. Endelig er de grønne og røde proteiner i dette sæt proteiner generelt de mest påviselige og bør derfor bruges til at mærke dine mindst rigelige proteiner, med de blå og infrarøde kanaler, der bruges til mere rigelige proteiner eller markeringsrum.
jeg håber, at dette kaster lys over flerfarvet billeddannelse med fluorescerende proteiner. Med det rigtige mikroskop og det rigtige valg af fluorescerende proteiner skal billeddannelse af fire farver samtidig være ret ligetil.
tak til vores gæsteblogger!
 Kurt Thorn er lektor ved UCSF, hvor han leder Nikon Imaging Center. Han modtog sin ph. d.i biofysik fra UCSF i laboratoriet i Ronald Vale, hvorefter han var stipendiat ved Bauer Center for Genomics Research ved Harvard University. Lær mere på hans lab hjemmeside eller hans mikroskopi blog.
Kurt Thorn er lektor ved UCSF, hvor han leder Nikon Imaging Center. Han modtog sin ph. d.i biofysik fra UCSF i laboratoriet i Ronald Vale, hvorefter han var stipendiat ved Bauer Center for Genomics Research ved Harvard University. Lær mere på hans lab hjemmeside eller hans mikroskopi blog.