Dieser Beitrag wurde von Kurt Thorn vom Nikon Imaging Center der UCSF verfasst.
Eine häufige Anforderung für Live-Cell-Imaging-Experimente ist die Fähigkeit, mehrere fluoreszenzmarkierte Spezies gleichzeitig zu verfolgen. Um dies mit fluoreszierenden Proteinmarkierungen zu tun, sind mehrere fluoreszierende Proteine erforderlich, deren Anregungs- und Emissionsspektren sich ausreichend unterscheiden, um sie in unterschiedlichen Fluoreszenzkanälen auf dem Mikroskop abzubilden. Mit der Verbreitung von fluoreszierenden Proteinen in den letzten Jahren gibt es viele fluoreszierende Proteinkombinationen, die zusammen abgebildet werden können, aber dies bedeutet auch, dass die Wahl der fluoreszierenden Proteine einige Überlegungen erfordert.

Der erste Schritt bei der Auswahl fluoreszierender Proteine für Ihr Mehrfarbenbildgebungsexperiment besteht darin, zu wissen, welche fluoreszierenden Proteine verfügbar sind. Da jeden Monat neue fluoreszierende Proteine veröffentlicht werden, ist die Entscheidung über das beste Protein für eine bestimmte Anwendung eine Herausforderung. Um Sie über die neuesten fluoreszierenden Proteine auf dem Laufenden zu halten, Ich pflege eine interaktive Grafik und Tabelle der besten derzeit verfügbaren fluoreszierenden Proteine.
Auswahl kompatibler fluoreszierender Proteine
Um einen Satz fluoreszierender Proteine auszuwählen, die zusammen abgebildet werden sollen, müssen Sie dieselben Faktoren berücksichtigen wie bei der Auswahl eines einzelnen fluoreszierenden Proteins (Helligkeit, Photostabilität usw.; weitere Informationen zu diesen Faktoren finden Sie im vorherigen Blogbeitrag). Darüber hinaus müssen Sie auch fluoreszierende Proteine auswählen, die voneinander unterschieden werden können und die mit der Optik der Mikroskope abgebildet werden können, die Sie verwenden möchten. Eine genaue Bestimmung, ob zwei fluoreszierende Proteine voneinander getrennt werden können, erfordert die Kenntnis ihrer Anregungs- und Emissionsspektren, aber eine gute Faustregel ist, dass sowohl die Peak-Anregungswellenlängen als auch die Peak-Emissionswellenlänge der beiden Proteine durch 50-60 nm getrennt sein sollten. Zum Beispiel können CFP (ex 430 nm / em 474 nm) und YFP (ex 514 nm / em 527 nm) zusammen abgebildet werden, aber CFP und GFP (ex 488 nm / em 507 nm) zeigen ein gewisses Übersprechen zwischen den beiden fluoreszierenden Proteinen. Wenn Sie fluoreszierende Proteine abbilden müssen, deren Spektren sich überlappen, gibt es Techniken wie das spektrale Entmischen, mit denen die fluoreszierenden Proteine getrennt werden können.
Sind Ihre fluoreszierenden Proteine mit Ihrer Mikroskopoptik kompatibel?
Um festzustellen, ob die fluoreszierenden Proteine, an denen Sie interessiert sind, mit Ihrer Mikroskopoptik kompatibel sind, sollten Sie die Anregungs- und Emissionsspektren Ihres Proteins mit den Filtersätzen oder Lasern Ihres Mikroskops vergleichen. Idealerweise möchten Sie eine wesentliche Überlappung zwischen den Anregungs- und Emissionsfiltern und den Anregungs- und Emissionsspektren des Proteins haben, damit das Protein von Ihrem Mikroskop gut angeregt wird und die Fluoreszenzemission des Proteins effizient vom Mikroskop erfasst wird. Um die Übereinstimmung zwischen einem fluoreszierenden Protein und einem Filtersatz zu vergleichen, bieten viele Filtersatzhersteller Werkzeuge zum Zeichnen der Fluoreszenzspektren von Proteinen und Farbstoffen sowie ihrer Filter an (siehe Chromas, Semrocks oder Omegas). Diese enthalten zwar nicht alle gängigen fluoreszierenden Proteine (insbesondere nicht die zuletzt veröffentlichten), können jedoch ein guter Ausgangspunkt sein. In vielen Fällen reicht es aus, ein Spektrum für ein eng verwandtes Protein zu verwenden, wenn Sie wissen, dass Ihr interessierendes Protein ein ähnliches Spektrum aufweist. Hier ist zum Beispiel ein Screenshot aus dem Chroma Spectra Viewer, der ein Standard-Cy3- oder Rhodamin-Filterset (Chroma # 49004) mit den Spektren von mCherry und TagRFP vergleicht.
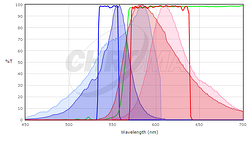
Hier ist das TagRFP-Spektrum in den dunkleren Farben und das mCherry-Spektrum in den helleren Farben dargestellt; Anregungsspektren sind blau und Emissionsspektren sind rot. Beides passt nicht perfekt zum Filtersatz, aber der Anregungsfilter regt mehr von der Spitze der TagRFP-Anregung an und der Emissionsfilter sammelt einen größeren Anteil der TagRFP-Emission als die mCherry-Emission. Für diesen Filtersatz würden wir erwarten, dass TagRFP ein helleres Signal liefert als mCherry. Im Allgemeinen funktionieren Filtersets, die für Rhodamin / Cy3 entwickelt wurden, mit rot fluoreszierenden Proteinen mit kürzerer Wellenlänge wie TagRFP oder mRuby2 besser als mit Proteinen mit längerer Wellenlänge wie mCherry. Hintergrundinformationen zu Fluoreszenz- und Filtersätzen finden Sie in der Vorlesung Einführung in die Fluoreszenzmikroskopie bei iBiology.
Häufig verwendete Filtersets & relevante fluoreszierende Proteine
Häufig verwendete Filtersets für die Mehrfarbenbildgebung umfassen solche, die für CFP, YFP und RFP ausgelegt sind, oder das Sedat Quad-Filterset, das für DAPI / Fluorescein / Rhodamin / Cy5 (z. B. Semrock) und die ähnliche 4-Laser-Kombination auf einem konfokalen405 / 488 / 561 / 640 nm). In unseren Händen sind die besten fluoreszierenden Proteine für die Bildgebung mit diesem Set mTagBFP2, EGFP oder eine der verbesserten GFP-Varianten, mRuby2 oder TagRFP-T, und ein Infrarot-fluoreszierendes Protein wie iFP1.4 oder iFP2.0. Beachten Sie, dass diese Infrarot-fluoreszierenden Proteine Biliverdin als Cofaktor benötigen und Sie daher möglicherweise Ihre Zellen mit Biliverdin ergänzen müssen, um eine maximale Helligkeit zu erzielen. In Säugetierzellen ist eine der verbesserten Faltungsvarianten von EGFP wie mEmerald oder Clover wahrscheinlich am besten; mNeonGreen ist ein noch neueres grün fluoreszierendes Protein, das extrem hell sein soll. In S. cerevisiae, wir haben getestet eine reihe von grün und rot fluoreszierende proteine mit diesem filter set und haben berichtet helligkeit messungen. Hier übertrifft EGFP die verbesserten Faltvarianten, vermutlich aufgrund der niedrigeren Wachstumstemperatur. Dies deutet jedoch auch darauf hin, dass es kein einzelnes fluoreszierendes Protein gibt, das für alle Organismen optimal ist, und dass Sie, wenn Sie das hellste Signal wünschen, möglicherweise mehrere Proteine in Ihrem interessierenden System ausprobieren müssen. Schließlich sind in diesem Satz von Proteinen die grünen und roten Proteine im Allgemeinen am besten nachweisbar und sollten daher verwendet werden, um Ihre am wenigsten vorhandenen Proteine zu markieren, wobei die blauen und infraroten Kanäle für häufigere Proteine oder Markierungskompartimente verwendet werden.
Ich hoffe, dies wirft ein Licht auf die mehrfarbige Bildgebung mit fluoreszierenden Proteinen. Mit dem richtigen Mikroskop und der richtigen Auswahl fluoreszierender Proteine sollte die gleichzeitige Abbildung von vier Farben ziemlich einfach sein.
Vielen Dank an unseren Gastblogger!
 Kurt Thorn ist Associate Professor an der UCSF, wo er das Nikon Imaging Center leitet. Er promovierte in Biophysik an der UCSF im Labor von Ronald Vale, danach war er Fellow am Bauer Center for Genomics Research der Harvard University. Erfahren Sie mehr auf seiner Labor-Webseite oder seinem Mikroskopie-Blog.
Kurt Thorn ist Associate Professor an der UCSF, wo er das Nikon Imaging Center leitet. Er promovierte in Biophysik an der UCSF im Labor von Ronald Vale, danach war er Fellow am Bauer Center for Genomics Research der Harvard University. Erfahren Sie mehr auf seiner Labor-Webseite oder seinem Mikroskopie-Blog.