Este post fue contribuido por Kurt Thorn del Centro de Imágenes Nikon en UCSF.
Un requisito común para los experimentos de imágenes de células vivas es la capacidad de seguir múltiples especies marcadas con fluorescencia simultáneamente. Para hacerlo con etiquetas de proteínas fluorescentes se requieren múltiples proteínas fluorescentes cuyos espectros de excitación y emisión difieren lo suficiente para que se puedan obtener imágenes en distintos canales fluorescentes en el microscopio. Con la proliferación de proteínas fluorescentes en los últimos años, hay muchas combinaciones de proteínas fluorescentes que se pueden visualizar juntas, pero esto también significa que la elección de proteínas fluorescentes requiere un poco de reflexión.

El primer paso para elegir proteínas fluorescentes para su experimento de imágenes multicolor es estar al tanto de las proteínas fluorescentes disponibles. Con nuevas proteínas fluorescentes que se publican cada mes, decidir cuál es la mejor proteína para una aplicación determinada es un desafío. Para ayudarlo a mantenerse al tanto de las últimas proteínas fluorescentes, mantengo un gráfico interactivo y una tabla de las mejores proteínas fluorescentes disponibles actualmente.
Elegir proteínas fluorescentes compatibles
Para elegir un conjunto de proteínas fluorescentes que se visualizarán juntas, deberá tener en cuenta los mismos factores que al elegir una proteína fluorescente individual (brillo, fotoestabilidad, etc.; consulte la publicación anterior del blog para obtener más información sobre estos factores). Además, también deberá elegir proteínas fluorescentes que se puedan distinguir entre sí y que se puedan obtener imágenes con la óptica del microscopio o los microscopios que pretenda usar. Una determinación precisa de si dos proteínas fluorescentes pueden separarse entre sí requiere el conocimiento de sus espectros de excitación y emisión, pero una buena regla general es que tanto las longitudes de onda de excitación de pico como la longitud de onda de emisión de pico de las dos proteínas deben estar separadas por 50-60 nm. Por ejemplo, el CFP (ex 430 nm / em 474 nm) y el YFP (ex 514 nm / em 527 nm) pueden visualizarse juntos, pero el CFP y el GFP (ex 488 nm / em 507 nm) muestran cierta interferencia entre las dos proteínas fluorescentes. Si debe obtener imágenes de proteínas fluorescentes cuyos espectros se superponen, hay técnicas, como la mezcla espectral, que se pueden usar para separar las proteínas fluorescentes, pero estas están más allá del alcance de este artículo.
¿Sus proteínas fluorescentes son compatibles con la óptica de su microscopio?
Para determinar si las proteínas fluorescentes que le interesan son compatibles con la óptica de su microscopio, querrá comparar los espectros de excitación y emisión de su proteína con los juegos de filtros o láseres de su microscopio. Idealmente, le gustaría tener una superposición sustancial entre los filtros de excitación y emisión y los espectros de excitación y emisión de la proteína, de modo que la proteína sea bien excitada por su microscopio y la emisión de fluorescencia de la proteína sea recogida de manera eficiente por el microscopio. Para comparar la coincidencia entre una proteína fluorescente y un conjunto de filtros, muchos proveedores de conjuntos de filtros proporcionan herramientas para trazar los espectros de fluorescencia de proteínas y colorantes y sus filtros (consulte Croma, Semrock u Omega). Si bien no contienen todas las proteínas fluorescentes de uso común (en particular, no las más recientes), pueden ser un buen punto de partida. En muchos casos, es suficiente usar un espectro para una proteína estrechamente relacionada, si sabe que su proteína de interés tiene un espectro similar. Por ejemplo, aquí hay una captura de pantalla del Visor de Espectros de Croma que compara un conjunto de filtros estándar Cy3 o Rodamina (Croma # 49004) con los espectros de mCherry y TagRFP.
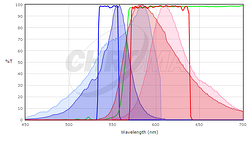
Aquí, el espectro TagRFP se muestra en los colores más oscuros y el espectro mCherry se muestra en los colores más claros; los espectros de excitación son azules y los espectros de emisión son rojos. Ninguno de los dos es una combinación perfecta con el conjunto de filtros, pero el filtro de excitación excita más del pico de la excitación TagRFP y el filtro de emisión recoge una fracción mayor de la emisión TagRFP que la emisión mCherry. Para este conjunto de filtros, esperaríamos que TagRFP diera una señal más brillante que mCherry. En general, los conjuntos de filtros diseñados para Rodamina / Cy3 funcionarán mejor con proteínas fluorescentes rojas de longitud de onda más corta como TagRFP o mRuby2 que con proteínas de longitud de onda más larga como mCherry. Para obtener información sobre conjuntos de fluorescencia y filtros, consulte la conferencia de Introducción a la microscopía de fluorescencia en iBiology.
Juegos de filtros de uso común & Proteínas fluorescentes relevantes
Los juegos de filtros de uso común para imágenes multicolores incluyen los diseñados para CFP, YFP y RFP o el juego de filtros cuádruples Sedat, diseñado para DAPI / Fluoresceína / Rodamina / Cy5 (por ejemplo, Semrock) y la combinación similar de 4 láseres en un sistema confocal (405 / 488 / 561 / 640 nm). En nuestras manos, las mejores proteínas fluorescentes para la obtención de imágenes con este conjunto son mTagBFP2, EGFP o una de las variantes mejoradas de GFP, mRuby2 o TagRFP-T, y una proteína fluorescente infrarroja como iFP1.4 o iFP2.0. Tenga en cuenta que estas proteínas fluorescentes infrarrojas requieren biliverdina como cofactor y, por lo tanto, es posible que deba complementar sus células con biliverdina para obtener el máximo brillo. En las células de mamíferos, una de las variantes de plegado mejoradas de EGFP como mEmerald o Trébol es probablemente la mejor; la mNeonGreen es una proteína fluorescente verde aún más nueva que se supone que es extremadamente brillante. En S. cerevisiae, hemos probado una serie de proteínas fluorescentes verdes y rojas con este conjunto de filtros y hemos reportado mediciones de brillo. Aquí, EGFP supera a las variantes de plegado mejoradas, presumiblemente debido a la temperatura de crecimiento más baja. Esto también sugiere, sin embargo, que no hay una sola proteína fluorescente óptima para todos los organismos y que si desea la señal más brillante, es posible que deba probar varias proteínas en su sistema de interés. Finalmente, en este conjunto de proteínas, las proteínas verdes y rojas son generalmente las más detectables y, por lo tanto, deben usarse para etiquetar las proteínas menos abundantes, con los canales azules e infrarrojos utilizados para proteínas más abundantes o compartimentos de marcado.
Espero que esto arroje algo de luz en imágenes multicolores con proteínas fluorescentes. Con el microscopio correcto y la elección correcta de proteínas fluorescentes, obtener imágenes de cuatro colores simultáneamente debería ser bastante sencillo.
¡Gracias a nuestro Blogger Invitado!
 Kurt Thorn es profesor asociado en UCSF, donde dirige el Centro de Imágenes Nikon. Recibió su doctorado en biofísica de la UCSF en el laboratorio de Ronald Vale, después de lo cual fue becario en el Centro Bauer de Investigación Genómica de la Universidad de Harvard. Obtenga más información en su página web de laboratorio o en su blog de microscopía.
Kurt Thorn es profesor asociado en UCSF, donde dirige el Centro de Imágenes Nikon. Recibió su doctorado en biofísica de la UCSF en el laboratorio de Ronald Vale, después de lo cual fue becario en el Centro Bauer de Investigación Genómica de la Universidad de Harvard. Obtenga más información en su página web de laboratorio o en su blog de microscopía.