Cet article a été rédigé par Kurt Thorn du Centre d’imagerie Nikon de l’UCSF.
Une exigence courante pour les expériences d’imagerie de cellules vivantes est la capacité de suivre simultanément plusieurs espèces marquées par fluorescence. Pour ce faire avec des étiquettes de protéines fluorescentes, il faut de multiples protéines fluorescentes dont les spectres d’excitation et d’émission diffèrent suffisamment pour qu’elles puissent être imagées dans des canaux fluorescents distincts au microscope. Avec la prolifération des protéines fluorescentes ces dernières années, de nombreuses combinaisons de protéines fluorescentes peuvent être imagées ensemble, mais cela signifie également que le choix des protéines fluorescentes nécessite une réflexion.

La première étape dans le choix des protéines fluorescentes pour votre expérience d’imagerie multicolore est de savoir quelles protéines fluorescentes sont disponibles. Avec de nouvelles protéines fluorescentes publiées chaque mois, décider de la meilleure protéine pour une application donnée est un défi. Pour vous tenir au courant des dernières protéines fluorescentes, je maintiens un graphique interactif et un tableau des meilleures protéines fluorescentes actuellement disponibles.
Choisir des protéines fluorescentes compatibles
Pour choisir un ensemble de protéines fluorescentes à imager ensemble, vous devrez tenir compte des mêmes facteurs que lors du choix d’une protéine fluorescente individuelle (luminosité, photostabilité, etc.; voir le billet de blog précédent pour plus de discussion sur ces facteurs). De plus, vous devrez également choisir des protéines fluorescentes qui peuvent être distinguées les unes des autres et qui peuvent être imagées avec l’optique du ou des microscopes que vous avez l’intention d’utiliser. Pour déterminer avec précision si deux protéines fluorescentes peuvent être séparées l’une de l’autre, il faut connaître leurs spectres d’excitation et d’émission, mais une bonne règle empirique est que les longueurs d’onde de crête d’excitation et la longueur d’onde de crête d’émission des deux protéines doivent être séparées de 50 à 60 nm. Par example, la PCP (ex 430 nm / em 474 nm) et la YFP (ex 514 nm/em 527 nm) peuvent être imagées ensemble mais la CFP et la GFP (ex 488 nm/em 507 nm) montrent une certaine diaphonie entre les deux protéines fluorescentes. Si vous devez imager des protéines fluorescentes dont les spectres se chevauchent, il existe des techniques, comme le démélange spectral, qui peuvent être utilisées pour séparer les protéines fluorescentes, mais celles-ci dépassent le cadre de cet article.
Vos protéines fluorescentes sont-elles compatibles avec l’optique de votre microscope ?
Pour déterminer si les protéines fluorescentes qui vous intéressent sont compatibles avec l’optique de votre microscope, vous voudrez comparer les spectres d’excitation et d’émission de votre protéine avec les jeux de filtres ou les lasers de votre microscope. Idéalement, vous souhaitez avoir un chevauchement substantiel entre les filtres d’excitation et d’émission et les spectres d’excitation et d’émission de la protéine, de sorte que la protéine soit bien excitée par votre microscope et que l’émission de fluorescence de la protéine soit efficacement collectée par le microscope. Pour comparer la correspondance entre une protéine fluorescente et un ensemble de filtres, de nombreux fournisseurs de filtres fournissent des outils pour tracer les spectres de fluorescence des protéines et des colorants et de leurs filtres (voir Chroma, Semrock ou Omega). Bien que celles-ci ne contiennent pas toutes les protéines fluorescentes d’usage courant (en particulier pas les plus récentes publiées), elles peuvent être un bon point de départ. Dans de nombreux cas, il suffit d’utiliser un spectre pour une protéine étroitement liée, si vous savez que votre protéine d’intérêt a un spectre similaire. Par exemple, voici une capture d’écran de la visionneuse de spectres Chroma comparant un ensemble de filtres Cy3 ou Rhodamine standard (Chroma #49004) aux spectres de mCherry et de TagRFP.
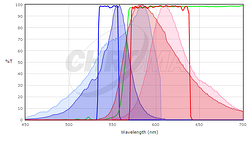
Ici, le spectre TagRFP est représenté dans les couleurs plus foncées et le spectre mCherry est représenté dans les couleurs plus claires; les spectres d’excitation sont bleus et les spectres d’émission sont rouges. Ni l’un ni l’autre ne correspond parfaitement à l’ensemble de filtres, mais le filtre d’excitation excite plus du pic de l’excitation de TagRFP et le filtre d’émission recueille une fraction plus importante de l’émission de TagRFP que l’émission de mCherry. Pour cet ensemble de filtres, nous nous attendons à ce que TagRFP donne un signal plus lumineux que mCherry. En général, les ensembles de filtres conçus pour la Rhodamine / Cy3 fonctionneront mieux avec des protéines fluorescentes rouges à longueur d’onde plus courte comme TagRFP ou mRuby2 que des protéines à longueur d’onde plus longue comme mCherry. Pour des informations générales sur la fluorescence et les ensembles de filtres, voir la conférence Introduction à la microscopie à fluorescence à iBiology.
Ensembles de filtres couramment utilisés & protéines fluorescentes pertinentes
Les ensembles de filtres couramment utilisés pour l’imagerie multicolore comprennent ceux conçus pour CFP, YFP et RFP ou l’ensemble de filtres Sedat Quad, conçu pour DAPI / Fluorescéine / Rhodamine / Cy5 (par exemple Semrock) et la combinaison similaire à 4 lasers sur un confocal (par exemple405 / 488 / 561 / 640 nm). Dans nos mains, les meilleures protéines fluorescentes pour l’imagerie avec cet ensemble sont mTagBFP2, EGFP ou l’une des variantes améliorées de GFP, mRuby2 ou TagRFP-T, et une protéine fluorescente infrarouge telle que iFP1.4 ou iFP2.0. Attention, ces protéines fluorescentes infrarouges nécessitent de la biliverdine comme cofacteur et vous devrez peut-être compléter vos cellules avec de la biliverdine pour une luminosité maximale. Dans les cellules de mammifères, l’une des variantes améliorées de pliage de l’EGFP comme mEmerald ou Clover est probablement la meilleure; le mNeonGreen est une protéine fluorescente verte encore plus récente censée être extrêmement brillante. En S. cerevisiae, nous avons testé un certain nombre de protéines fluorescentes vertes et rouges avec cet ensemble de filtres et avons rapporté des mesures de luminosité. Ici, EGFP surpasse les variantes de pliage améliorées, probablement en raison de la température de croissance plus basse. Cela suggère également, cependant, qu’il n’y a pas de protéine fluorescente unique optimale pour tous les organismes et que si vous voulez le signal le plus brillant, vous devrez peut-être essayer plusieurs protéines dans votre système d’intérêt. Enfin, dans cet ensemble de protéines, les protéines vertes et rouges sont généralement les plus détectables et doivent donc être utilisées pour marquer vos protéines les moins abondantes, avec les canaux bleus et infrarouges utilisés pour les protéines les plus abondantes ou les compartiments de marquage.
J’espère que cela éclaire l’imagerie multicolore avec des protéines fluorescentes. Avec le bon microscope et le bon choix de protéines fluorescentes, l’imagerie simultanée de quatre couleurs devrait être assez simple.
Merci à notre Blogueur invité!
 Kurt Thorn est professeur associé à l’UCSF, où il dirige le Centre d’imagerie Nikon. Il a obtenu son doctorat en biophysique de l’UCSF dans le laboratoire de Ronald Vale, après quoi il a été chercheur au Bauer Center for Genomics Research de l’Université Harvard. Pour en savoir plus, consultez la page Web de son laboratoire ou son blog sur la microscopie.
Kurt Thorn est professeur associé à l’UCSF, où il dirige le Centre d’imagerie Nikon. Il a obtenu son doctorat en biophysique de l’UCSF dans le laboratoire de Ronald Vale, après quoi il a été chercheur au Bauer Center for Genomics Research de l’Université Harvard. Pour en savoir plus, consultez la page Web de son laboratoire ou son blog sur la microscopie.