detta inlägg bidrog av Kurt Thorn från Nikon Imaging Center vid UCSF.
ett vanligt krav för levande cellbildningsexperiment är förmågan att följa flera fluorescerande märkta arter samtidigt. För att göra det med fluorescerande proteinetiketter krävs flera fluorescerande proteiner vars exciterings-och emissionsspektra skiljer sig tillräckligt för att de ska avbildas i distinkta fluorescerande kanaler på mikroskopet. Med spridningen av fluorescerande proteiner under de senaste åren finns det många fluorescerande proteinkombinationer som kan avbildas tillsammans, men det betyder också att valet av fluorescerande proteiner kräver viss tanke.

det första steget i att välja fluorescerande proteiner för ditt flerfärgsbildningsexperiment är att vara medveten om vilka fluorescerande proteiner som finns tillgängliga. Med nya fluorescerande proteiner som publiceras varje månad är det en utmaning att bestämma det bästa proteinet för en given applikation. För att hålla dig uppdaterad om de senaste fluorescerande proteinerna, upprätthåller jag en interaktiv graf och tabell över de bästa fluorescerande proteinerna som för närvarande finns tillgängliga.
välja kompatibla fluorescerande proteiner
för att välja en uppsättning fluorescerande proteiner som ska avbildas tillsammans måste du överväga samma faktorer som när du väljer ett individuellt fluorescerande protein (ljusstyrka, fotostabilitet och så vidare; Se föregående blogginlägg för mer diskussion om dessa faktorer). Dessutom måste du också välja fluorescerande proteiner som kan särskiljas från varandra och som kan avbildas med optiken på mikroskopet / mikroskopen du tänker använda. En noggrann bestämning av huruvida två fluorescerande proteiner kan separeras från varandra kräver kunskap om deras exciterings-och emissionsspektra, men en bra tumregel är att både de maximala exciteringsvåglängderna och toppemissionsvåglängden för de två proteinerna ska separeras med 50-60 nm. Till exempel kan CFP (ex 430 nm / em 474 nm) och YFP (ex 514 nm / em 527 nm) avbildas tillsammans men CFP och GFP (ex 488 nm / em 507 nm) visar en viss överhörning mellan de två fluorescerande proteinerna. Om du måste avbilda fluorescerande proteiner vars spektra överlappar varandra finns det tekniker, som spektral blandning, som kan användas för att separera de fluorescerande proteinerna, men dessa ligger utanför ramen för detta inlägg.
är dina fluorescerande proteiner kompatibla med din mikroskopoptik?
för att avgöra om de fluorescerande proteinerna du är intresserad av är kompatibla med din mikroskopop, vill du jämföra excitations-och emissionsspektra för ditt protein med filteruppsättningarna eller lasrarna på ditt mikroskop. Helst skulle du vilja ha väsentlig överlappning mellan excitations-och emissionsfiltret och excitations-och emissionsspektra för proteinet, så att proteinet är väl upphetsat av ditt mikroskop och fluorescensutsläppet av proteinet samlas effektivt upp av mikroskopet. För att jämföra matchningen mellan ett fluorescerande protein och en filteruppsättning tillhandahåller många filteruppsättningsleverantörer verktyg för att plotta fluorescensspektra av proteiner och färgämnen och deras filter (Se krom, Semrock eller Omega). Även om dessa inte innehåller alla fluorescerande proteiner i vanligt bruk (särskilt inte de senast publicerade), kan de vara en bra utgångspunkt. I många fall är det tillräckligt att använda ett spektrum för ett närbesläktat protein, om du vet att ditt protein av intresse har ett liknande spektrum. Till exempel, här är en skärmdump från Chroma Spectra Viewer jämföra en standard Cy3 eller Rhodamine filter set (Chroma #49004) till spektra av både mCherry och TagRFP.
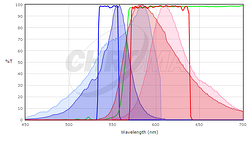
Här visas TagRFP-spektrumet i de mörkare färgerna och mCherry-spektrumet visas i de ljusare färgerna; excitationsspektra är blå och emissionsspektra är röda. Inte heller är en perfekt matchning till filteruppsättningen men exciteringsfiltret exciterar mer av toppen av TagRFP-excitationen och emissionsfiltret samlar in en större bråkdel av TagRFP-emissionen än mcherry-emissionen. För denna filteruppsättning förväntar vi oss att TagRFP ger en ljusare signal än mCherry. I allmänhet kommer filteruppsättningar utformade för Rhodamin / Cy3 att fungera bättre med kortare våglängdsröda fluorescerande proteiner som TagRFP eller mRuby2 än längre våglängdsproteiner som mCherry. För bakgrund om fluorescens-och filteruppsättningar, se Introduktion till Fluorescensmikroskopiföreläsning vid iBiology.
vanliga filtersatser & relevanta fluorescerande proteiner
vanliga filtersatser för multicolor imaging inkluderar de som är utformade för CFP, YFP och RFP eller Sedat Quad filter set, designad för DAPI / Fluorescein / Rhodamine / Cy5 (t. ex. Semrock ’ s) och den liknande 4-laserkombinationen på en konfokal (405 / 488 / 561 / 640 nm). I våra händer är de bästa fluorescerande proteinerna för avbildning med denna uppsättning mTagBFP2, EGFP eller en av de förbättrade GFP-varianterna, mRuby2 eller TagRFP-T, och ett infrarött fluorescerande protein som iFP1.4 eller iFP2.0. Akta dig för att dessa infraröda fluorescerande proteiner kräver biliverdin som en kofaktor och så kan du behöva komplettera dina celler med biliverdin för maximal ljusstyrka. I däggdjursceller är en av de förbättrade vikningsvarianterna av EGFP som mEmerald eller Clover förmodligen bäst; mNeonGreen är ett ännu nyare grönt fluorescerande protein som ska vara extremt ljust. I S. cerevisiae, vi har testat ett antal gröna och röda fluorescerande proteiner med denna filteruppsättning och har rapporterat ljusmätningar. Här överträffar EGFP de förbättrade vikningsvarianterna, förmodligen på grund av den lägre tillväxttemperaturen. Detta tyder dock också på att det inte finns något enda fluorescerande protein optimalt för alla organismer och att om du vill ha den ljusaste signalen kan du behöva prova flera proteiner i ditt system av intresse. Slutligen, i denna uppsättning proteiner är de gröna och röda proteinerna i allmänhet de mest detekterbara och bör därför användas för att märka dina minst rikliga proteiner, med de blå och infraröda kanalerna som används för mer rikliga proteiner eller märkningsfack.
jag hoppas att detta kastar lite ljus på multicolor imaging med fluorescerande proteiner. Med rätt mikroskop och rätt val av fluorescerande proteiner bör avbilda Fyra färger samtidigt vara ganska enkelt.
tack till vår Gästbloggare!
 Kurt Thorn är docent vid UCSF, där han leder Nikon Imaging Center. Han fick sin doktorsexamen i biofysik från UCSF i laboratoriet i Ronald Vale, varefter han var stipendiat vid Bauer Center for Genomics Research vid Harvard University. Läs mer på hans lab webbsida eller hans mikroskopi blogg.
Kurt Thorn är docent vid UCSF, där han leder Nikon Imaging Center. Han fick sin doktorsexamen i biofysik från UCSF i laboratoriet i Ronald Vale, varefter han var stipendiat vid Bauer Center for Genomics Research vid Harvard University. Läs mer på hans lab webbsida eller hans mikroskopi blogg.