este post foi contribuído por Kurt Thorn do centro de imagens Nikon na UCSF.
uma exigência comum para experiências de imagiologia de células vivas é a capacidade de seguir várias espécies marcadas com fluorescência simultaneamente. Para fazê-lo com rótulos de proteínas fluorescentes requer várias proteínas fluorescentes cuja excitação e espectros de emissão diferem suficientemente para que elas sejam fotografadas em canais fluorescentes distintos no microscópio. Com a proliferação de proteínas fluorescentes nos últimos anos, existem muitas combinações de proteínas fluorescentes que podem ser visualizadas em conjunto, mas isso também significa que a escolha de proteínas fluorescentes requer algum pensamento.

o primeiro passo na escolha de proteínas fluorescentes para o seu experimento de imagem multi-cores é estar ciente do que as proteínas fluorescentes estão disponíveis. Com novas proteínas fluorescentes sendo publicadas todos os meses, decidir sobre a melhor proteína para uma determinada aplicação é um desafio. Para ajudar a mantê-lo a par das mais recentes proteínas fluorescentes, eu manter um gráfico interativo e tabela das melhores proteínas fluorescentes atualmente disponíveis.
Escolher compatível com proteínas fluorescentes
Para escolher um conjunto de proteínas fluorescentes para ser trabalhada em conjunto, você precisará considerar os mesmos fatores que, quando a escolha de um indivíduo proteína fluorescente (luz, fotoestabilidade, e assim por diante; veja o post anterior para uma discussão mais desses fatores). Além disso, você também terá que escolher as proteínas fluorescentes que podem ser distinguidas umas das outras e que podem ser fotografadas com a óptica no(s) microscópio (s) que você pretende usar. Uma determinação precisa de se duas proteínas fluorescentes podem ser separadas umas das outras requer o conhecimento de sua excitação e espectros de emissão, mas uma boa regra é que tanto os comprimentos de onda de excitação de pico e comprimento de onda de emissão de pico das duas proteínas devem ser separados por 50-60 nm. Por exemplo, o PFR (ex 430 nm / em 474 nm) e o YFP (ex 514 nm / em 527 nm) podem ser visualizados em conjunto, mas o PFR e o GFP (ex 488 nm / em 507 nm) mostram algum crosstalk entre as duas proteínas fluorescentes. Se você deve imagem de proteínas fluorescentes cujos espectros se sobrepõem, há técnicas, como unmixing espectral, que podem ser usadas para separar as proteínas fluorescentes, mas estas estão além do escopo deste post.As suas proteínas fluorescentes são compatíveis com o microscópio óptico?
para determinar se as proteínas fluorescentes em que está interessado são compatíveis com a sua óptica microscópica, você vai querer comparar os espectros de excitação e emissão da sua proteína com os conjuntos de filtros ou lasers no seu microscópio. Idealmente, você gostaria de ter uma sobreposição substancial entre os filtros de excitação e emissão e os espectros de excitação e emissão da proteína, de modo que a proteína está bem excitada pelo seu microscópio e a emissão de fluorescência da proteína é eficientemente coletada pelo microscópio. Para comparar a correspondência entre uma proteína fluorescente e um conjunto de filtros, muitos vendedores de filtros fornecem ferramentas para plotar os espectros de fluorescência de proteínas e corantes e seus filtros (ver Chroma, Semrock, ou Omega). Embora estes não contenham todas as proteínas fluorescentes em uso comum (particularmente não as mais recentemente publicadas), eles podem ser um bom ponto de partida. Em muitos casos, é suficiente usar um espectro para uma proteína intimamente relacionada, se você sabe que a sua proteína de interesse tem um espectro semelhante. Por exemplo, aqui está uma imagem do espectador de espectros cromados comparando um conjunto de filtros Cy3 ou Rhodamine padrão (Chroma #49004) com os espectros de ambos mCherry e TagRFP.
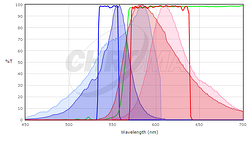
aqui, o espectro de TagRFP é mostrado nas cores mais escuras e o espectro de mCherry é mostrado nas cores mais claras; espectros de excitação são azuis e espectros de emissão são vermelhos. Também não é uma combinação perfeita com o conjunto de filtros, mas o filtro de excitação excita mais do Pico da excitação TagRFP e o filtro de emissão coleta uma fração maior da emissão TagRFP do que a emissão mCherry. Para este conjunto de filtros, nós esperaríamos TagRFP para dar um sinal mais brilhante do que mCherry. Em geral, os conjuntos de filtros projetados para Rhodamine / Cy3 funcionarão melhor com as proteínas fluorescentes vermelho de comprimento de onda mais curto como TagRFP ou mRuby2 do que as proteínas de comprimento de onda mais longo como mCherry. For background on fluorescence and filter sets, see the Introduction to Fluorescence Microscopy lecture at iBiology.
Comumente utilizados conjuntos de filtro & relevante proteínas fluorescentes
Comumente utilizados conjuntos de filtro para multicolor de imagem incluem aqueles projetados para o PCP, YFP, e RFP ou o Sedat Quad conjunto de filtro, projetado para DAPI / Fluoresceína / Rodamina / Cy5 (e.g. Semrock) e semelhantes, de 4 de laser de combinação em um confocal (405 / 488 / 561 / 640 nm). Em nossas mãos as melhores proteínas fluorescentes para imagens com este conjunto são mTagBFP2, EGFP ou uma das variantes melhor GFP, mRuby2 ou TagRFP-T, e uma proteína fluorescente infravermelha como iFP1.4 ou iFP2.0. Cuidado com o facto de estas proteínas fluorescentes infravermelhas necessitarem de biliverdina como cofactor, pelo que poderá necessitar de completar as suas células com biliverdin para obter o brilho máximo. Em células de mamíferos, uma das variantes de dobragem melhorada do EGFP como mEmerald ou trevo é provavelmente melhor; o rneongreen é uma proteína verde fluorescente ainda mais recente que supostamente é extremamente brilhante. Em S. cerevisiae, testamos uma série de proteínas fluorescentes verdes e vermelhas com este conjunto de filtros e relatamos medições de brilho. Aqui, o EGFP supera as variantes dobráveis melhoradas, presumivelmente devido à menor temperatura de crescimento. Isto também sugere, no entanto, que não há uma única proteína fluorescente ideal para todos os organismos e que se você quiser o sinal mais brilhante, você pode precisar de tentar várias proteínas em seu sistema de interesse. Finalmente, neste conjunto de proteínas, as proteínas verde e vermelha são geralmente as mais detectáveis e por isso devem ser usadas para marcar as suas proteínas menos abundantes, com os canais azul e infravermelho usados para proteínas mais abundantes ou compartimentos de marcação.
espero que isto produza alguma luz em imagens multicolor com proteínas fluorescentes. Com o microscópio direito e a escolha certa de proteínas fluorescentes, imagens de quatro cores simultaneamente deve ser bastante simples.Obrigado ao nosso blogueiro convidado!
 Kurt Thorn é um professor associado na UCSF, onde dirige o centro de imagens Nikon. Ele recebeu seu doutorado em biofísica da UCSF no laboratório de Ronald Vale, após o qual ele foi um fellow no Bauer Center for Genomics Research na Universidade de Harvard. Saiba mais na página web do laboratório ou no blog de microscopia.
Kurt Thorn é um professor associado na UCSF, onde dirige o centro de imagens Nikon. Ele recebeu seu doutorado em biofísica da UCSF no laboratório de Ronald Vale, após o qual ele foi um fellow no Bauer Center for Genomics Research na Universidade de Harvard. Saiba mais na página web do laboratório ou no blog de microscopia.