Questo post è stato contribuito da Kurt Thorn del Nikon Imaging Center presso UCSF.
Un requisito comune per gli esperimenti di imaging cellulare vivo è la capacità di seguire più specie fluorescenti etichettate contemporaneamente. Per fare ciò con le etichette fluorescenti della proteina richiede le proteine fluorescenti multiple di cui gli spettri dell’emissione e di eccitazione differiscono sufficientemente per loro essere imaged nei canali fluorescenti distinti sul microscopio. Con la proliferazione di proteine fluorescenti negli ultimi anni, ci sono molte combinazioni di proteine fluorescenti che possono essere imaged insieme, ma questo significa anche che la scelta di proteine fluorescenti richiede qualche pensiero.

Il primo passo nella scelta di proteine fluorescenti per il vostro esperimento di imaging multi-colore è quello di essere consapevoli di ciò che le proteine fluorescenti sono disponibili. Con nuove proteine fluorescenti pubblicati ogni mese, decidere la migliore proteina per una data applicazione è una sfida. Per aiutarti a tenerti al passo con le ultime proteine fluorescenti, mantengo un grafico interattivo e una tabella delle migliori proteine fluorescenti attualmente disponibili.
Scelta di proteine fluorescenti compatibili
Per scegliere un insieme di proteine fluorescenti da fotografare insieme, è necessario considerare gli stessi fattori di quando si sceglie una singola proteina fluorescente (luminosità, fotostabilità e così via; vedere il post del blog precedente per ulteriori discussioni su questi fattori). Inoltre, sarà anche necessario scegliere proteine fluorescenti che possono essere distinte l’una dall’altra e che possono essere imaged con l’ottica sul microscopio(s) che si intende utilizzare. Una determinazione accurata se due proteine fluorescenti possono essere separate l’una dall’altra richiede la conoscenza dei loro spettri di eccitazione ed emissione, ma una buona regola empirica è che sia le lunghezze d’onda di eccitazione di picco che la lunghezza d’onda di emissione di picco delle due proteine dovrebbero essere separate da 50-60 nm. Ad esempio, CFP (ex 430 nm / em 474 nm) e YFP (ex 514 nm / em 527 nm) possono essere imaged insieme, ma CFP e GFP (ex 488 nm / em 507 nm) mostrano una certa diafonia tra le due proteine fluorescenti. Se è necessario immagine proteine fluorescenti cui spettri si sovrappongono, ci sono tecniche, come spettrale unmixing, che possono essere utilizzati per separare le proteine fluorescenti, ma questi sono oltre lo scopo di questo post.
Le proteine fluorescenti sono compatibili con l’ottica del microscopio?
Per determinare se le proteine fluorescenti che ti interessano sono compatibili con l’ottica del tuo microscopio, dovrai confrontare gli spettri di eccitazione ed emissione della tua proteina con i set di filtri o i laser sul tuo microscopio. Idealmente, si desidera avere una sostanziale sovrapposizione tra i filtri di eccitazione ed emissione e gli spettri di eccitazione ed emissione della proteina, in modo che la proteina sia ben eccitata dal microscopio e l’emissione di fluorescenza della proteina sia efficacemente raccolta dal microscopio. Per confrontare la corrispondenza tra una proteina fluorescente e un set di filtri, molti fornitori di set di filtri forniscono strumenti per tracciare gli spettri di fluorescenza di proteine e coloranti e i loro filtri (vedere Chroma, Semrock o Omega). Sebbene questi non contengano tutte le proteine fluorescenti di uso comune (in particolare non quelle pubblicate più di recente), possono essere un buon punto di partenza. In molti casi è sufficiente utilizzare uno spettro per una proteina strettamente correlata, se sai che la tua proteina di interesse ha uno spettro simile. Ad esempio, ecco uno screenshot dal Chroma Spectra Viewer che confronta un set di filtri Cy3 o Rhodamine standard (Chroma #49004) con gli spettri di mCherry e TagRFP.
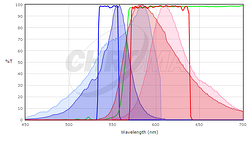
Qui, lo spettro TagRFP è mostrato nei colori più scuri e lo spettro mCherry è mostrato nei colori più chiari; gli spettri di eccitazione sono blu e gli spettri di emissione sono rossi. Nessuno dei due è una corrispondenza perfetta al set di filtri, ma il filtro di eccitazione eccita più del picco dell’eccitazione TagRFP e il filtro di emissione raccoglie una frazione maggiore dell’emissione TagRFP rispetto all’emissione mCherry. Per questo set di filtri, ci aspetteremmo che TagRFP dia un segnale più luminoso di mCherry. In generale, i set di filtri progettati per rodamina / Cy3 funzioneranno meglio con proteine fluorescenti rosse a lunghezza d’onda più corta come TagRFP o mRuby2 rispetto alle proteine a lunghezza d’onda più lunga come mCherry. Per informazioni sulla fluorescenza e sui set di filtri, vedere la lezione di Introduzione alla microscopia a fluorescenza presso iBiology.
Set di filtri comunemente usati &proteine fluorescenti rilevanti
I set di filtri comunemente usati per l’imaging multicolore includono quelli progettati per CFP, YFP e RFP o il set di filtri Sedat Quad, progettato per DAPI / Fluoresceina / rodamina / Cy5 (ad esempio Semrock) e la simile combinazione a 4 laser su un (405 / 488 / 561 / 640 nm). Nelle nostre mani le migliori proteine fluorescenti per l’imaging con questo set sono mTagBFP2, EGFP o una delle varianti GFP migliorate, mRuby2 o TagRFP-T e una proteina fluorescente a infrarossi come iFP1.4 o iFP2.0. Attenzione che queste proteine fluorescenti a infrarossi richiedono biliverdina come cofattore e quindi potrebbe essere necessario integrare le cellule con biliverdina per la massima luminosità. Nelle cellule di mammifero, una delle varianti pieghevoli migliorate di EGFP come mEmerald o Clover è probabilmente la migliore; mNeonGreen è una proteina fluorescente verde ancora più recente che dovrebbe essere estremamente luminosa. In S. cerevisiae, abbiamo testato un certo numero di proteine fluorescenti verdi e rosse con questo set di filtri e abbiamo riportato misurazioni della luminosità. Qui, EGFP supera le varianti di piegatura migliorate, presumibilmente a causa della temperatura di crescita più bassa. Questo suggerisce anche, tuttavia, che non esiste una singola proteina fluorescente ottimale per tutti gli organismi e che se si desidera il segnale più luminoso, potrebbe essere necessario provare diverse proteine nel sistema di interesse. Infine, in questo insieme di proteine le proteine verdi e rosse sono generalmente le più rilevabili e quindi dovrebbero essere utilizzate per etichettare le proteine meno abbondanti, con i canali blu e infrarossi utilizzati per proteine più abbondanti o scomparti di marcatura.
Spero che questo faccia luce sull’imaging multicolore con proteine fluorescenti. Con il microscopio giusto e la giusta scelta di proteine fluorescenti, l’imaging di quattro colori contemporaneamente dovrebbe essere piuttosto semplice.
Grazie al nostro Guest Blogger!
 Kurt Thorn è professore associato presso UCSF, dove dirige il Nikon Imaging Center. Ha conseguito il dottorato di ricerca in biofisica presso l’UCSF nel laboratorio di Ronald Vale, dopo di che è stato fellow presso il Bauer Center for Genomics Research presso l’Università di Harvard. Scopri di più nella sua pagina web di laboratorio o nel suo blog di microscopia.
Kurt Thorn è professore associato presso UCSF, dove dirige il Nikon Imaging Center. Ha conseguito il dottorato di ricerca in biofisica presso l’UCSF nel laboratorio di Ronald Vale, dopo di che è stato fellow presso il Bauer Center for Genomics Research presso l’Università di Harvard. Scopri di più nella sua pagina web di laboratorio o nel suo blog di microscopia.