Die Bioinformatik umfasst die Integration von Computern, Softwaretools und Datenbanken, um biologische Fragen zu beantworten. Bioinformatische Ansätze werden häufig für große Initiativen verwendet, die große Datensätze generieren. Zwei wichtige groß angelegte Aktivitäten, die Bioinformatik verwenden, sind Genomik und Proteomik. Genomik bezieht sich auf die Analyse von Genomen. Ein Genom kann als vollständiger Satz von DNA-Sequenzen betrachtet werden, die für das Erbmaterial kodieren, das von Generation zu Generation weitergegeben wird. Diese DNA-Sequenzen umfassen alle Gene (die funktionelle und physikalische Einheit der Vererbung, die von den Eltern an die Nachkommen weitergegeben wird) und Transkripte (die RNA-Kopien, die den ersten Schritt bei der Entschlüsselung der genetischen Information darstellen), die im Genom enthalten sind. Daher bezieht sich Genomik auf die Sequenzierung und Analyse all dieser genomischen Entitäten, einschließlich Gene und Transkripte, in einem Organismus. Proteomik hingegen bezieht sich auf die Analyse des vollständigen Satzes von Proteinen oder Proteomen. Neben der Genomik und Proteomik gibt es viele weitere Bereiche der Biologie, in denen die Bioinformatik angewendet wird (i.e., Metabolomik, Transkriptomik). Jeder dieser wichtigen Bereiche der Bioinformatik zielt darauf ab, komplexe biologische Systeme zu verstehen.
Viele Wissenschaftler bezeichnen die nächste Welle der Bioinformatik heute als Systembiologie, einen Ansatz zur Bewältigung neuer und komplexer biologischer Fragen. Die Systembiologie umfasst die Integration von Genomik-, Proteomik- und Bioinformatikinformationen, um eine Gesamtsystemansicht einer biologischen Entität zu erstellen.
Zum Beispiel, wie ein Signalweg in einer Zelle funktioniert, kann durch Systembiologie angesprochen werden. Die Gene, die an dem Signalweg beteiligt sind, wie sie interagieren und wie Modifikationen die Ergebnisse stromabwärts verändern, können alle mithilfe der Systembiologie modelliert werden. Jedes System, in dem die Informationen digital dargestellt werden können, bietet eine potenzielle Anwendung für die Bioinformatik. So kann Bioinformatik von einzelnen Zellen auf ganze Ökosysteme angewendet werden. Durch das Verständnis der vollständigen „Stücklisten“ in einem Genom gewinnen Wissenschaftler ein besseres Verständnis komplexer biologischer Systeme. Das Verständnis der Wechselwirkungen, die zwischen all diesen Teilen in einem Genom oder Proteom auftreten, stellt die nächste Komplexitätsstufe im System dar. Durch diese Ansätze hat die Bioinformatik das Potenzial, wichtige Einblicke in unser Verständnis und unsere Modellierung zu geben, wie sich bestimmte menschliche Krankheiten oder gesunde Zustände manifestieren.
Die Anfänge der Bioinformatik gehen auf Margaret Dayhoff im Jahr 1968 und ihre Sammlung von Proteinsequenzen zurück, die als Atlas der Proteinsequenz und -struktur bekannt ist. Eines der ersten bedeutenden Experimente in der Bioinformatik war die Anwendung eines Sequenzähnlichkeitssuchprogramms zur Identifizierung der Ursprünge eines viralen Gens. In dieser Studie verwendeten die Wissenschaftler eines der ersten Computerprogramme zur Suche nach Sequenzähnlichkeit (FASTP), um festzustellen, dass der Inhalt von v-sis, einer krebserregenden Virussequenz, dem gut charakterisierten zellulären PDGF-Gen am ähnlichsten war. Dieses überraschende Ergebnis lieferte wichtige mechanistische Erkenntnisse für Biologen, die daran arbeiten, wie diese Virussequenz Krebs verursacht. Von dieser ersten Anwendung von Computern in der Biologie ist das Gebiet der Bioinformatik explodiert. Das Wachstum der Bioinformatik verläuft parallel zur Entwicklung der DNA-Sequenzierungstechnologie. So wie die Entwicklung des Mikroskops in den späten 1600er Jahren die Biowissenschaften revolutionierte, indem Anton Van Leeuwenhoek zum ersten Mal Zellen betrachten konnte, hat die DNA-Sequenzierungstechnologie das Gebiet der Bioinformatik revolutioniert. Das schnelle Wachstum der Bioinformatik kann durch das Wachstum von DNA-Sequenzen veranschaulicht werden, die im öffentlichen Repository für Nukleotidsequenzen namens GenBank enthalten sind.
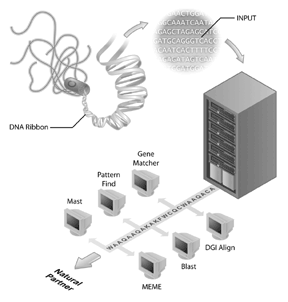
Genomsequenzierungsprojekte sind zu den Flaggschiffen vieler Bioinformatik-Initiativen geworden. Das Humangenomsequenzierungsprojekt ist ein Beispiel für ein erfolgreiches Genomsequenzierungsprojekt, aber auch viele andere Genome wurden sequenziert und werden sequenziert. In der Tat waren die ersten Genome, die sequenziert wurden, von Viren (d.h., der Phage MS2) und Bakterien, wobei das Genom von Haemophilus influenzae Rd das erste Genom eines frei lebenden Organismus ist, das in den öffentlichen Sequenzdatenbanken hinterlegt ist. Diese Leistung wurde mit weniger Fanfare aufgenommen als die Fertigstellung des menschlichen Genoms, aber es wird deutlich, dass die Sequenzierung anderer Genome heute ein wichtiger Schritt für die Bioinformatik ist. Die Genomsequenz selbst hat jedoch nur begrenzte Informationen. Um genomische Informationen zu interpretieren, muss eine vergleichende Analyse von Sequenzen durchgeführt werden, und ein wichtiges Reagenz für diese Analysen sind die öffentlich zugänglichen Sequenzdatenbanken. Ohne die Datenbanken von Sequenzen (wie GenBank), in denen Biologen Informationen über ihre Sequenz von Interesse erfasst haben, wäre ein Großteil der reichhaltigen Informationen aus Genomsequenzierungsprojekten nicht verfügbar.
So wie Entwicklungen in der Mikroskopie Entdeckungen in der Zellbiologie vorwegnahmen, lassen neue Entdeckungen in der Informationstechnologie und Molekularbiologie Entdeckungen in der Bioinformatik vorwegnehmen. In der Tat ist ein wichtiger Teil des Bereichs der Bioinformatik die Entwicklung neuer Technologien, die es der Wissenschaft der Bioinformatik ermöglichen, sehr schnell voranzukommen. Auf der Computerseite haben das Internet, neue Softwareentwicklungen, neue Algorithmen und die Entwicklung der Computerclustertechnologie der Bioinformatik große Sprünge in Bezug auf die Datenmenge ermöglicht, die effizient analysiert werden kann. Auf der Laborseite haben sich neue Technologien und Methoden wie DNA-Sequenzierung, serielle Analyse der Genexpression (SAGE), Microarrays und neue Massenspektrometriechemikalien in einem ebenso rasanten Tempo entwickelt, so dass Wissenschaftler Daten für Analysen mit einer unglaublichen Geschwindigkeit produzieren können. Die Bioinformatik bietet sowohl die Plattformtechnologien, die es Wissenschaftlern ermöglichen, mit den großen Datenmengen umzugehen, die durch Genomik- und Proteomikinitiativen erzeugt werden, als auch den Ansatz, diese Daten zu interpretieren. In vielerlei Hinsicht bietet die Bioinformatik die Werkzeuge für die Anwendung wissenschaftlicher Methoden auf groß angelegte Daten und sollte als wissenschaftlicher Ansatz für die Beantwortung vieler neuer und unterschiedlicher Arten biologischer Fragen angesehen werden.
Das Wort Bioinformatik ist in der Wissenschaft zu einem sehr beliebten „Buzz“ -Wort geworden. Viele Wissenschaftler finden Bioinformatik spannend, weil sie das Potenzial birgt, in eine ganz neue Welt von Neuland einzutauchen. Bioinformatik ist eine neue Wissenschaft und eine neue Denkweise, die möglicherweise zu vielen relevanten biologischen Entdeckungen führen könnte. Obwohl die Technologie die Bioinformatik ermöglicht, geht es in der Bioinformatik immer noch sehr stark um Biologie. Biologische Fragen treiben alle bioinformatischen Experimente an. Wichtige biologische Fragen können von der Bioinformatik beantwortet werden und umfassen das Verständnis der Genotyp-Phänotyp-Verbindung für menschliche Krankheiten, das Verständnis von Struktur-Funktions-Beziehungen für Proteine und das Verständnis biologischer Netzwerke. Bioinformatiker stellen häufig fest, dass die Reagenzien, die zur Beantwortung dieser interessanten biologischen Fragen erforderlich sind, nicht existieren. Daher besteht ein großer Teil der Arbeit eines Bioinformatikers darin, Werkzeuge und Technologien als Teil des Fragestellungsprozesses zu entwickeln. Für viele ist die Bioinformatik sehr beliebt, da Wissenschaftler sowohl ihre Biologie- als auch ihre Computerkenntnisse auf die Entwicklung von Reagenzien für die Bioinformatikforschung anwenden können. Viele Wissenschaftler stellen fest, dass die Bioinformatik ein aufregendes Neuland der wissenschaftlichen Forschung mit großem Potenzial für die menschliche Gesundheit und die Gesellschaft darstellt.
Die Zukunft der Bioinformatik ist die Integration. Zum Beispiel wird die Integration einer Vielzahl von Datenquellen wie klinische und genomische Daten es uns ermöglichen, Krankheitssymptome zu verwenden, um genetische Mutationen vorherzusagen und umgekehrt. Die Integration von GIS-Daten wie Karten und Wettersystemen mit Daten zur Pflanzengesundheit und zum Genotyp ermöglicht es uns, erfolgreiche Ergebnisse landwirtschaftlicher Experimente vorherzusagen. Ein weiteres zukünftiges Forschungsgebiet der Bioinformatik ist die groß angelegte vergleichende Genomik. Zum Beispiel wird die Entwicklung von Werkzeugen, die 10-Wege-Vergleiche von Genomen durchführen können, die Entdeckungsrate in diesem Bereich der Bioinformatik vorantreiben. In diesem Sinne könnte die Modellierung und Visualisierung vollständiger Netzwerke komplexer Systeme in Zukunft verwendet werden, um vorherzusagen, wie das System (oder die Zelle) beispielsweise auf ein Medikament reagiert. Eine technische Reihe von Herausforderungen steht der Bioinformatik gegenüber und wird durch schnellere Computer, technologische Fortschritte im Plattenspeicherplatz und erhöhte Bandbreite angegangen, aber bei weitem eine der größten Hürden für die Bioinformatik heute ist die geringe Anzahl von Forschern auf dem Gebiet. Dies ändert sich, da die Bioinformatik an die Spitze der Forschung rückt, aber diese Verzögerung des Fachwissens hat zu echten Wissenslücken über Bioinformatik in der Forschungsgemeinschaft geführt. Schließlich wird eine zentrale Forschungsfrage für die Zukunft der Bioinformatik sein, wie komplexe biologische Beobachtungen wie Genexpressionsmuster und Proteinnetzwerke rechnerisch verglichen werden können. In der Bioinformatik geht es darum, biologische Beobachtungen in ein Modell umzuwandeln, das ein Computer versteht. Dies ist eine sehr herausfordernde Aufgabe, da die Biologie sehr komplex sein kann. Dieses Problem, wie phänotypische Daten wie Verhalten, Elektrokardiogramme und Pflanzengesundheit in eine computerlesbare Form digitalisiert werden können, bietet spannende Herausforderungen für zukünftige Bioinformatiker.
(Dieser Artikel basiert auf einem Interview mit Francis Ouellette, Direktor des UBC Bioinformatics Centre)