La bioinformatica comporta l’integrazione di computer, strumenti software e database nel tentativo di affrontare questioni biologiche. Approcci bioinformatici sono spesso utilizzati per le principali iniziative che generano grandi insiemi di dati. Due importanti attività su larga scala che utilizzano la bioinformatica sono la genomica e la proteomica. La genomica si riferisce all’analisi dei genomi. Un genoma può essere pensato come l’insieme completo di sequenze di DNA che codifica per il materiale ereditario che viene trasmesso di generazione in generazione. Queste sequenze di DNA includono tutti i geni (l’unità funzionale e fisica dell’eredità passata dal genitore alla prole) e i trascritti (le copie di RNA che sono il passo iniziale nella decodifica delle informazioni genetiche) inclusi nel genoma. Pertanto, la genomica si riferisce al sequenziamento e all’analisi di tutte queste entità genomiche, inclusi geni e trascritti, in un organismo. La proteomica, d’altra parte, si riferisce all’analisi del set completo di proteine o proteomi. Oltre alla genomica e proteomica, ci sono molte altre aree della biologia in cui viene applicata la bioinformatica (io.e., metabolomica, trascrittomica). Ognuna di queste importanti aree in bioinformatica mira a comprendere sistemi biologici complessi.
Molti scienziati oggi si riferiscono alla prossima ondata in bioinformatica come biologia dei sistemi, un approccio per affrontare nuove e complesse questioni biologiche. La biologia dei sistemi comporta l’integrazione di genomica, proteomica e informazioni bioinformatiche per creare una visione dell’intero sistema di un’entità biologica.
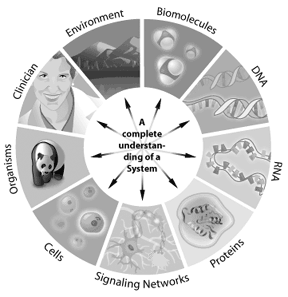
Ad esempio, come funziona un percorso di segnalazione in una cellula può essere affrontato attraverso la biologia dei sistemi. I geni coinvolti nel percorso, come interagiscono e come le modifiche modificano i risultati a valle, possono essere modellati utilizzando la biologia dei sistemi. Qualsiasi sistema in cui le informazioni possono essere rappresentate digitalmente offre una potenziale applicazione per la bioinformatica. Quindi la bioinformatica può essere applicata da singole cellule a interi ecosistemi. Comprendendo le “liste delle parti” complete in un genoma, gli scienziati stanno guadagnando una migliore comprensione dei sistemi biologici complessi. Comprendere le interazioni che si verificano tra tutte queste parti in un genoma o proteoma rappresenta il prossimo livello di complessità nel sistema. Attraverso questi approcci, la bioinformatica ha il potenziale per offrire intuizioni chiave nella nostra comprensione e modellazione di come si manifestano specifiche malattie umane o stati sani.
L’inizio della bioinformatica può essere fatta risalire a Margaret Dayhoff nel 1968 e alla sua collezione di sequenze proteiche conosciute come Atlas of Protein Sequence and Structure. Uno dei primi esperimenti significativi in bioinformatica è stata l’applicazione di un programma di ricerca di somiglianza sequenza per l’identificazione delle origini di un gene virale. In questo studio, gli scienziati hanno utilizzato uno dei primi programmi per computer di ricerca di similarità di sequenza (chiamato FASTP), per determinare che il contenuto di v-sis, una sequenza virale che causa il cancro, era più simile al gene PDGF cellulare ben caratterizzato. Questo risultato sorprendente ha fornito importanti intuizioni meccanicistiche per i biologi che lavorano su come questa sequenza virale provoca il cancro. Da questa prima applicazione iniziale dei computer alla biologia, il campo della bioinformatica è esploso. La crescita della bioinformatica è parallela allo sviluppo della tecnologia di sequenziamento del DNA. Allo stesso modo in cui lo sviluppo del microscopio alla fine del 1600 rivoluzionò le scienze biologiche consentendo ad Anton Van Leeuwenhoek di guardare le cellule per la prima volta, la tecnologia di sequenziamento del DNA ha rivoluzionato il campo della bioinformatica. La rapida crescita della bioinformatica può essere illustrata dalla crescita di sequenze di DNA contenute nel repository pubblico di sequenze nucleotidiche chiamate GenBank.
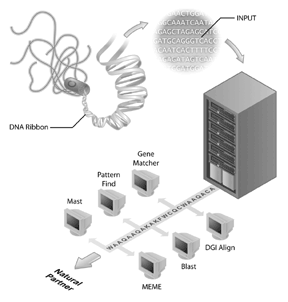
I progetti di sequenziamento del genoma sono diventati i fiori all’occhiello di molte iniziative di bioinformatica. Il progetto di sequenziamento del genoma umano è un esempio di un progetto di sequenziamento del genoma di successo, ma molti altri genomi sono stati sequenziati e vengono sequenziati. Infatti, i primi genomi da sequenziare erano di virus (cioè, il fago MS2) e batteri, con il genoma di Haemophilus influenzae Rd essendo il primo genoma di un organismo vivente libero ad essere depositato nelle banche dati di sequenza pubblica. Questo risultato è stato ricevuto con meno clamore rispetto al completamento del genoma umano, ma sta diventando chiaro che il sequenziamento di altri genomi è un passo importante per la bioinformatica oggi. Tuttavia, la sequenza del genoma di per sé ha informazioni limitate. Per interpretare le informazioni genomiche, è necessario eseguire un’analisi comparativa delle sequenze e un reagente importante per queste analisi sono i database di sequenze accessibili al pubblico. Senza i database di sequenze (come GenBank), in cui i biologi hanno catturato informazioni sulla loro sequenza di interesse, gran parte delle ricche informazioni ottenute da progetti di sequenziamento del genoma non sarebbe disponibile.
Allo stesso modo in cui gli sviluppi in microscopia prefiguravano scoperte in biologia cellulare, nuove scoperte in tecnologia dell’informazione e biologia molecolare stanno prefigurando scoperte in bioinformatica. In effetti, una parte importante del campo della bioinformatica è lo sviluppo di nuove tecnologie che consentono alla scienza della bioinformatica di procedere ad un ritmo molto veloce. Dal punto di vista informatico, Internet, nuovi sviluppi software, nuovi algoritmi e lo sviluppo della tecnologia dei cluster di computer hanno permesso alla bioinformatica di fare grandi passi avanti in termini di quantità di dati che possono essere analizzati in modo efficiente. Sul lato del laboratorio, nuove tecnologie e metodi come il sequenziamento del DNA, l’analisi seriale dell’espressione genica (SAGE), i microarray e le nuove chimiche di spettrometria di massa si sono sviluppati a un ritmo altrettanto vescicante consentendo agli scienziati di produrre dati per le analisi a un ritmo incredibile. Bioinformatica fornisce sia le tecnologie di piattaforma che consentono agli scienziati di affrontare le grandi quantità di dati prodotti attraverso iniziative di genomica e proteomica, nonché l’approccio per interpretare questi dati. In molti modi, la bioinformatica fornisce gli strumenti per applicare il metodo scientifico a dati su larga scala e dovrebbe essere vista come un approccio scientifico per porre molti nuovi e diversi tipi di domande biologiche.
La parola bioinformatica è diventata una parola “buzz” molto popolare nella scienza. Molti scienziati trovano bioinformatica eccitante perché detiene il potenziale per immergersi in un intero nuovo mondo di territorio inesplorato. La bioinformatica è una nuova scienza e un nuovo modo di pensare che potrebbe potenzialmente portare a molte scoperte biologiche rilevanti. Sebbene la tecnologia consenta la bioinformatica, la bioinformatica riguarda ancora molto la biologia. Le domande biologiche guidano tutti gli esperimenti di bioinformatica. Importanti questioni biologiche possono essere affrontate dalla bioinformatica e comprendono la comprensione della connessione genotipo-fenotipo per la malattia umana, la comprensione delle relazioni struttura-funzione per le proteine e la comprensione delle reti biologiche. I bioinformatici trovano spesso che i reagenti necessari per rispondere a queste interessanti domande biologiche non esistono. Pertanto, gran parte del lavoro di un bioinformatico è la costruzione di strumenti e tecnologie come parte del processo di porre la domanda. Per molti, la bioinformatica è molto popolare perché gli scienziati possono applicare sia la loro biologia che le loro abilità informatiche allo sviluppo di reagenti per la ricerca bioinformatica. Molti scienziati stanno scoprendo che la bioinformatica è un nuovo entusiasmante territorio di interrogativi scientifici con un grande potenziale a beneficio della salute umana e della società.
Il futuro della bioinformatica è l’integrazione. Ad esempio, l’integrazione di un’ampia varietà di fonti di dati come i dati clinici e genomici ci consentirà di utilizzare i sintomi della malattia per prevedere le mutazioni genetiche e viceversa. L’integrazione di dati GIS, come mappe, sistemi meteorologici, con dati sulla salute delle colture e sul genotipo, ci consentirà di prevedere i risultati positivi degli esperimenti agricoli. Un’altra futura area di ricerca in bioinformatica è la genomica comparativa su larga scala. Ad esempio, lo sviluppo di strumenti che possono fare confronti a 10 vie di genomi spingerà in avanti il tasso di scoperta in questo campo della bioinformatica. Lungo queste linee, la modellazione e la visualizzazione di reti complete di sistemi complessi potrebbero essere utilizzati in futuro per prevedere come il sistema (o cellula) reagisce, ad un farmaco, per esempio. Un insieme tecnico di sfide affronta bioinformatica e viene affrontato da computer più veloci, progressi tecnologici nello spazio di archiviazione su disco, e una maggiore larghezza di banda, ma di gran lunga uno dei più grandi ostacoli che affrontano bioinformatica oggi, è il piccolo numero di ricercatori nel campo. Questo sta cambiando come bioinformatica si muove in prima linea della ricerca, ma questo ritardo nella competenza ha portato a lacune reali nella conoscenza della bioinformatica nella comunità di ricerca. Infine, una domanda chiave di ricerca per il futuro della bioinformatica sarà come confrontare computazionalmente osservazioni biologiche complesse, come i modelli di espressione genica e le reti proteiche. La bioinformatica consiste nel convertire le osservazioni biologiche in un modello che un computer capirà. Questo è un compito molto impegnativo dal momento che la biologia può essere molto complessa. Questo problema di come digitalizzare i dati fenotipici come il comportamento, gli elettrocardiogrammi e la salute delle colture in una forma leggibile dal computer offre sfide entusiasmanti per i futuri bioinformatici.
(Questo articolo è basato su un’intervista con Francis Ouellette, direttore del Centro di bioinformatica UBC)