La bioinformática implica la integración de computadoras, herramientas de software y bases de datos en un esfuerzo por abordar cuestiones biológicas. Los enfoques bioinformáticos se utilizan a menudo para iniciativas importantes que generan grandes conjuntos de datos. Dos actividades importantes a gran escala que utilizan bioinformática son la genómica y la proteómica. La genómica se refiere al análisis de los genomas. Un genoma puede considerarse como el conjunto completo de secuencias de ADN que codifica el material hereditario que se transmite de generación en generación. Estas secuencias de ADN incluyen todos los genes (la unidad funcional y física de la herencia transmitida de padres a hijos) y transcripciones (las copias de ARN que son el paso inicial para decodificar la información genética) incluidos en el genoma. Por lo tanto, la genómica se refiere a la secuenciación y el análisis de todas estas entidades genómicas, incluidos los genes y las transcripciones, en un organismo. La proteómica, por otro lado, se refiere al análisis del conjunto completo de proteínas o proteomas. Además de la genómica y la proteómica, hay muchas más áreas de la biología en las que se está aplicando la bioinformática (i.e., metabolómica, transcriptómica). Cada una de estas áreas importantes en bioinformática tiene como objetivo comprender sistemas biológicos complejos.
Hoy en día, muchos científicos se refieren a la próxima ola de bioinformática como biología de sistemas, un enfoque para abordar cuestiones biológicas nuevas y complejas. La biología de sistemas implica la integración de información genómica, proteómica y bioinformática para crear una visión de sistema completa de una entidad biológica.
Por ejemplo, cómo funciona una vía de señalización en una célula se puede abordar a través de la biología de sistemas. Los genes involucrados en la vía, cómo interactúan y cómo las modificaciones cambian los resultados aguas abajo, pueden modelarse utilizando la biología de sistemas. Cualquier sistema en el que la información se pueda representar digitalmente ofrece una aplicación potencial para la bioinformática. Por lo tanto, la bioinformática se puede aplicar desde células individuales a ecosistemas enteros. Al comprender las » listas de partes «completas de un genoma, los científicos están adquiriendo una mejor comprensión de los sistemas biológicos complejos. Comprender las interacciones que ocurren entre todas estas partes de un genoma o proteoma representa el siguiente nivel de complejidad en el sistema. A través de estos enfoques, la bioinformática tiene el potencial de ofrecer información clave sobre nuestra comprensión y modelos de cómo se manifiestan enfermedades humanas específicas o estados saludables.
El comienzo de la bioinformática se remonta a Margaret Dayhoff en 1968 y su colección de secuencias de proteínas conocida como el Atlas de la Secuencia y Estructura de Proteínas. Uno de los primeros experimentos significativos en bioinformática fue la aplicación de un programa de búsqueda de similitud de secuencias para la identificación de los orígenes de un gen viral. En este estudio, los científicos utilizaron uno de los primeros programas informáticos de búsqueda de similitud de secuencias (llamado FASTP) para determinar que el contenido de v-sis, una secuencia viral que causa cáncer, era más similar al gen celular bien caracterizado PDGF. Este sorprendente resultado proporcionó importantes conocimientos mecanicistas para los biólogos que trabajan sobre cómo esta secuencia viral causa cáncer. A partir de esta primera aplicación inicial de las computadoras a la biología, el campo de la bioinformática ha explotado. El crecimiento de la bioinformática es paralelo al desarrollo de la tecnología de secuenciación de ADN. De la misma manera que el desarrollo del microscopio a finales de los años 1600 revolucionó las ciencias biológicas al permitir a Anton Van Leeuwenhoek observar las células por primera vez, la tecnología de secuenciación de ADN ha revolucionado el campo de la bioinformática. El rápido crecimiento de la bioinformática puede ser ilustrado por el crecimiento de secuencias de ADN contenidas en el repositorio público de secuencias de nucleótidos llamado GenBank.
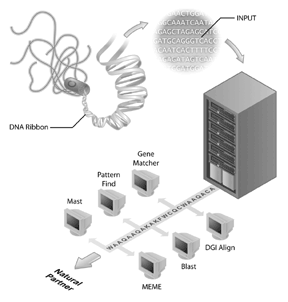
Los proyectos de secuenciación del genoma se han convertido en los buques insignia de muchas iniciativas de bioinformática. El proyecto de secuenciación del genoma humano es un ejemplo de un proyecto exitoso de secuenciación del genoma, pero muchos otros genomas también se han secuenciado y se están secuenciando. De hecho, los primeros genomas que se secuenciaron fueron de virus (i. e., el fago MS2) y bacterias, siendo el genoma de Haemophilus influenzae Rd el primer genoma de un organismo vivo libre que se deposita en los bancos de datos públicos de secuencias. Este logro se recibió con menos fanfarria que la finalización del genoma humano, pero cada vez está más claro que la secuenciación de otros genomas es un paso importante para la bioinformática de hoy en día. Sin embargo, la secuencia del genoma por sí sola tiene información limitada. Para interpretar la información genómica, es necesario realizar análisis comparativos de secuencias y un reactivo importante para estos análisis son las bases de datos de secuencias de acceso público. Sin las bases de datos de secuencias (como GenBank), en las que los biólogos han capturado información sobre su secuencia de interés, gran parte de la rica información obtenida de los proyectos de secuenciación del genoma no estaría disponible.
De la misma manera que los avances en microscopía presagiaban descubrimientos en biología celular, los nuevos descubrimientos en tecnología de la información y biología molecular presagian descubrimientos en bioinformática. De hecho, una parte importante del campo de la bioinformática es el desarrollo de nuevas tecnologías que permiten que la ciencia de la bioinformática avance a un ritmo muy rápido. En el lado de la computadora, Internet, los nuevos desarrollos de software, los nuevos algoritmos y el desarrollo de la tecnología de clúster de computadoras han permitido que la bioinformática dé grandes saltos en términos de la cantidad de datos que se pueden analizar de manera eficiente. En el lado del laboratorio, las nuevas tecnologías y métodos, como la secuenciación de ADN, el análisis en serie de expresión génica (SAGE), los microarrays y las nuevas químicas de espectrometría de masas, se han desarrollado a un ritmo igualmente vertiginoso, lo que permite a los científicos producir datos para análisis a un ritmo increíble. La bioinformática proporciona las tecnologías de plataforma que permiten a los científicos lidiar con las grandes cantidades de datos producidos a través de iniciativas de genómica y proteómica, así como el enfoque para interpretar estos datos. En muchos sentidos, la bioinformática proporciona las herramientas para aplicar métodos científicos a datos a gran escala y debe verse como un enfoque científico para hacer muchos tipos nuevos y diferentes de preguntas biológicas.
La palabra bioinformática se ha convertido en una palabra muy popular en la ciencia. Muchos científicos consideran que la bioinformática es emocionante porque tiene el potencial de sumergirse en un mundo completamente nuevo de territorio inexplorado. La bioinformática es una nueva ciencia y una nueva forma de pensar que podría conducir a muchos descubrimientos biológicos relevantes. Aunque la tecnología permite la bioinformática, la bioinformática sigue siendo una cuestión de biología. Las preguntas biológicas impulsan todos los experimentos bioinformáticos. La bioinformática puede abordar importantes cuestiones biológicas, entre las que se incluyen la comprensión de la conexión genotipo-fenotipo de la enfermedad humana, la comprensión de las relaciones entre la estructura y la función de las proteínas y la comprensión de las redes biológicas. Los bioinformáticos a menudo encuentran que los reactivos necesarios para responder a estas interesantes preguntas biológicas no existen. Por lo tanto, una gran parte del trabajo de un bioinformático es construir herramientas y tecnologías como parte del proceso de hacer la pregunta. Para muchos, la bioinformática es muy popular porque los científicos pueden aplicar tanto su biología como sus habilidades informáticas al desarrollo de reactivos para la investigación bioinformática. Muchos científicos están descubriendo que la bioinformática es un nuevo y emocionante territorio de cuestionamiento científico con un gran potencial para beneficiar la salud humana y la sociedad.
El futuro de la bioinformática es la integración. Por ejemplo, la integración de una amplia variedad de fuentes de datos, como datos clínicos y genómicos, nos permitirá utilizar los síntomas de la enfermedad para predecir mutaciones genéticas y viceversa. La integración de datos SIG, como mapas y sistemas meteorológicos, con datos de salud de los cultivos y genotipos, nos permitirá predecir los resultados satisfactorios de los experimentos agrícolas. Otra futura área de investigación en bioinformática es la genómica comparativa a gran escala. Por ejemplo, el desarrollo de herramientas que puedan hacer comparaciones de 10 direcciones de genomas impulsará la tasa de descubrimiento en este campo de la bioinformática. En este sentido, el modelado y la visualización de redes completas de sistemas complejos podrían utilizarse en el futuro para predecir cómo reacciona el sistema (o la célula), por ejemplo, a un fármaco. Un conjunto de desafíos técnicos se enfrenta a la bioinformática y se está abordando con computadoras más rápidas, avances tecnológicos en el espacio de almacenamiento en disco y un mayor ancho de banda, pero uno de los mayores obstáculos que enfrenta la bioinformática en la actualidad es el pequeño número de investigadores en el campo. Esto está cambiando a medida que la bioinformática se mueve a la vanguardia de la investigación, pero este retraso en la experiencia ha dado lugar a lagunas reales en el conocimiento de la bioinformática en la comunidad de investigación. Finalmente, una pregunta clave de investigación para el futuro de la bioinformática será cómo comparar computacionalmente observaciones biológicas complejas, como patrones de expresión génica y redes de proteínas. La bioinformática consiste en convertir las observaciones biológicas en un modelo que una computadora pueda entender. Esta es una tarea muy desafiante, ya que la biología puede ser muy compleja. Este problema de cómo digitalizar datos fenotípicos como el comportamiento, los electrocardiogramas y la salud de los cultivos en una forma legible por computadora ofrece desafíos emocionantes para los bioinformáticos futuros.
(Este artículo se basa en una entrevista con Francis Ouellette, Director del Centro de Bioinformática de la UBC)