Bioinformatyka obejmuje integrację komputerów, narzędzi programowych i baz danych w celu rozwiązania problemów biologicznych. Podejścia bioinformatyczne są często stosowane w przypadku dużych inicjatyw generujących duże zbiory danych. Dwie ważne działania na dużą skalę, które wykorzystują bioinformatykę, to genomika i proteomika. Genomika odnosi się do analizy genomów. Genom może być uważany za kompletny zestaw sekwencji DNA, które kodują materiał dziedziczny, który jest przekazywany z pokolenia na pokolenie. Te sekwencje DNA obejmują wszystkie geny (funkcjonalną i fizyczną jednostkę dziedziczności przekazywaną z rodzica na potomstwo) i transkrypty (kopie RNA, które są początkowym etapem dekodowania informacji genetycznej) zawarte w genomie. Tak więc genomika odnosi się do sekwencjonowania i analizy wszystkich tych jednostek genomowych, w tym genów i transkryptów, w organizmie. Proteomika, z drugiej strony, odnosi sie do analizy kompletnego zestawu bialek lub proteomu. Oprócz genomiki i proteomiki, istnieje wiele innych dziedzin biologii, w których stosuje się bioinformatykę (m.in.e., metabolomika, transkryptomika). Każdy z tych ważnych obszarów bioinformatyki ma na celu zrozumienie złożonych systemów biologicznych.
wielu naukowców odnosi się dzisiaj do następnej fali bioinformatyki jako biologii systemowej, podejścia do rozwiązywania nowych i złożonych problemów biologicznych. Biologia systemowa obejmuje integrację genomiki, proteomiki i informacji bioinformatycznych w celu stworzenia całego widoku systemu jednostki biologicznej.
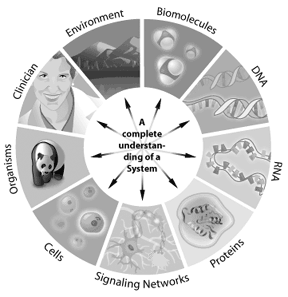
na przykład, jak działa szlak sygnałowy w komórce, można rozwiązać za pomocą biologii systemowej. Geny zaangażowane w Szlak, sposób ich interakcji i jak modyfikacje zmieniają wyniki w dalszym ciągu, mogą być modelowane za pomocą biologii systemowej. Każdy system, w którym Informacje mogą być reprezentowane cyfrowo, oferuje potencjalne zastosowanie dla bioinformatyki. W ten sposób bioinformatykę można zastosować od pojedynczych komórek do całych ekosystemów. Dzięki zrozumieniu kompletnych „list części” w genomie, naukowcy zyskują lepsze zrozumienie złożonych systemów biologicznych. Zrozumienie interakcji zachodzących między wszystkimi tymi częściami w genomie lub proteomie stanowi kolejny poziom złożoności systemu. Dzięki tym podejściom bioinformatyka ma potencjał, aby zaoferować kluczowe wgląd w nasze zrozumienie i modelowanie tego, jak objawiają się konkretne choroby ludzkie lub zdrowe Stany.
początki bioinformatyki sięgają Margaret Dayhoff w 1968 roku i jej kolekcji sekwencji białkowych znanych jako Atlas sekwencji i struktury białek. Jednym z wczesnych znaczących eksperymentów w bioinformatyce było zastosowanie programu wyszukiwania podobieństwa sekwencji do identyfikacji pochodzenia genu wirusowego. W tym badaniu naukowcy użyli jednego z pierwszych programów komputerowych do wyszukiwania podobieństwa sekwencji (zwanego FASTP), aby ustalić, że zawartość V-sis, rakotwórczej sekwencji wirusowej, była najbardziej podobna do dobrze scharakteryzowanego komórkowego genu PDGF. Ten zaskakujący wynik dostarczył ważnych mechanistycznych spostrzeżeń dla biologów pracujących nad tym, jak ta sekwencja wirusa powoduje raka. Od pierwszego wstępnego zastosowania komputerów do biologii eksplodowała dziedzina bioinformatyki. Rozwój Bioinformatyki jest równoległy do rozwoju technologii sekwencjonowania DNA. W ten sam sposób, w jaki rozwój mikroskopu pod koniec 1600 roku zrewolucjonizował nauki biologiczne, umożliwiając Antonowi Van Leeuwenhoekowi spojrzenie na komórki po raz pierwszy, technologia sekwencjonowania DNA zrewolucjonizowała dziedzinę bioinformatyki. Szybki wzrost bioinformatyki może być zilustrowany przez wzrost sekwencji DNA zawartych w publicznym repozytorium sekwencji nukleotydowych zwanych GenBank.
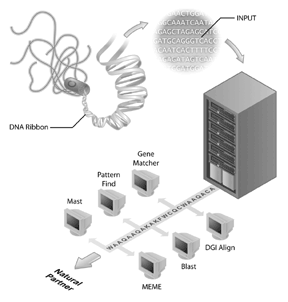
projekty sekwencjonowania genomu stały się sztandarami wielu inicjatyw bioinformatycznych. Projekt sekwencjonowania ludzkiego genomu jest przykładem udanego projektu sekwencjonowania genomu, ale wiele innych genomów zostało również zsekwencjonowanych i są sekwencjonowane. W rzeczywistości pierwszymi genomami do zsekwencjonowania były wirusy (tj., FAG MS2) i bakterii, przy czym Genom Haemophilus influenzae Rd jest pierwszym genomem wolnego żywego organizmu, który ma zostać zdeponowany w publicznych bazach danych sekwencji. To osiągnięcie zostało przyjęte z mniejszą pompą niż Ukończenie ludzkiego genomu, ale staje się jasne, że sekwencjonowanie innych genomów jest ważnym krokiem dla bioinformatyki dzisiaj. Jednak sekwencja genomu sama w sobie ma ograniczone informacje. Aby zinterpretować informacje genomowe, należy przeprowadzić analizę porównawczą sekwencji, a ważnym odczynnikiem do tych analiz są publicznie dostępne bazy sekwencji. Bez baz danych sekwencji (takich jak GenBank), w których biolodzy uchwycili informacje o ich sekwencji zainteresowania, wiele bogatych informacji uzyskanych z projektów sekwencjonowania genomu nie byłoby dostępnych.
w ten sam sposób rozwój mikroskopii zapowiadał odkrycia w biologii komórki, nowe odkrycia w technologii informacyjnej i biologii molekularnej zapowiadały odkrycia w bioinformatyce. W rzeczywistości ważną częścią dziedziny Bioinformatyki jest rozwój nowej technologii, która umożliwia naukę bioinformatyki postępować w bardzo szybkim tempie. Po stronie komputera, Internet, Nowe oprogramowanie, nowe algorytmy i rozwój technologii klastrów komputerowych umożliwiły bioinformatyce wielki skok pod względem ilości danych, które mogą być skutecznie analizowane. Od strony laboratoryjnej nowe technologie i metody, takie jak sekwencjonowanie DNA, seryjna analiza ekspresji genów (SAGE), mikromacierze i nowe chemikalia spektrometrii masowej, rozwinęły się w równie szybkim tempie, umożliwiając naukowcom tworzenie danych do analiz w niesamowitym tempie. Bioinformatyka zapewnia zarówno technologie platformowe, które umożliwiają naukowcom radzenie sobie z dużymi ilościami danych wytwarzanych przez inicjatywy genomiki i proteomiki, jak i podejście do interpretacji tych danych. Pod wieloma względami bioinformatyka zapewnia narzędzia do stosowania metody naukowej do danych na dużą skalę i powinna być postrzegana jako naukowe podejście do zadawania wielu nowych i różnych rodzajów pytań biologicznych.
słowo bioinformatyka stało się bardzo popularnym słowem „buzz” w nauce. Wielu naukowców uważa bioinformatykę za ekscytującą, ponieważ ma potencjał, aby zanurzyć się w zupełnie nowym świecie niezbadanego terytorium. Bioinformatyka jest nową nauką i nowym sposobem myślenia, który może potencjalnie prowadzić do wielu istotnych odkryć biologicznych. Chociaż technologia umożliwia bioinformatykę, bioinformatyka jest nadal bardzo dużo o biologii. Pytania biologiczne napędzają wszystkie eksperymenty bioinformatyczne. Ważne pytania biologiczne mogą być rozwiązywane przez bioinformatyki i obejmują zrozumienie genotypu-fenotyp związku dla choroby ludzkiej, zrozumienie struktury do funkcji relacji dla białek i zrozumienie sieci biologicznych. Bioinformatycy często uważają, że odczynniki niezbędne do odpowiedzi na te interesujące pytania biologiczne nie istnieją. Tak więc dużą częścią pracy bioinformatyka jest budowanie narzędzi i technologii w ramach procesu zadawania pytania. Dla wielu bioinformatyka jest bardzo popularna, ponieważ naukowcy mogą zastosować zarówno swoją biologię, jak i umiejętności komputerowe do opracowywania odczynników do badań bioinformatycznych. Wielu naukowców odkrywa, że bioinformatyka jest ekscytującym nowym obszarem badań naukowych z ogromnym potencjałem, aby przynieść korzyści zdrowiu ludzkiemu i społeczeństwu.
przyszłością Bioinformatyki jest integracja. Na przykład integracja szerokiej gamy źródeł danych, takich jak dane kliniczne i genomowe, pozwoli nam wykorzystać objawy choroby do przewidywania mutacji genetycznych i odwrotnie. Integracja danych GIS, takich jak mapy, systemy pogodowe, z danymi dotyczącymi zdrowia upraw i genotypu, pozwoli nam przewidywać pomyślne wyniki eksperymentów rolniczych. Inną przyszłą dziedziną badań bioinformatycznych jest genomika porównawcza na dużą skalę. Na przykład, rozwój narzędzi, które mogą robić 10-way porównania genomów popchnie do przodu szybkość odkrywania w tej dziedzinie bioinformatyki. W związku z tym modelowanie i wizualizacja pełnych sieci złożonych systemów może być wykorzystana w przyszłości do przewidywania, jak system (lub komórka) reaguje, na przykład na lek. Techniczny zestaw wyzwań stojących przed bioinformatyką i jest rozwiązywany przez szybsze Komputery, postęp technologiczny w przestrzeni dyskowej i zwiększoną przepustowość, ale zdecydowanie jednym z największych przeszkód stojących dziś przed bioinformatyką jest niewielka liczba naukowców w tej dziedzinie. Zmienia się to, ponieważ bioinformatyka przesuwa się na pierwszy plan badań, ale ta opóźniona wiedza specjalistyczna doprowadziła do rzeczywistych luk w wiedzy na temat bioinformatyki w środowisku badawczym. Wreszcie, kluczowym pytaniem badawczym dla przyszłości bioinformatyki będzie jak obliczeniowo porównywać złożone obserwacje biologiczne, takie jak wzorce ekspresji genów i sieci białkowe. Bioinformatyka polega na przekształcaniu obserwacji biologicznych w model, który komputer zrozumie. Jest to bardzo trudne zadanie, ponieważ Biologia może być bardzo złożona. Ten problem digitalizacji danych fenotypowych, takich jak zachowanie, elektrokardiogramy i zdrowie upraw w formie czytelnej dla komputera, stanowi ekscytujące wyzwanie dla przyszłych bioinformatyków.
(artykuł powstał na podstawie wywiadu z Francisem Ouellette, Dyrektorem Centrum Bioinformatyki UBC)