bioinformatik innebär integration av datorer, mjukvaruverktyg och databaser i ett försök att ta itu med biologiska frågor. Bioinformatik används ofta för stora initiativ som genererar stora datamängder. Två viktiga storskaliga aktiviteter som använder bioinformatik är genomik och proteomik. Genomik hänvisar till analysen av genom. Ett Genom kan betraktas som den kompletta uppsättningen DNA-sekvenser som kodar för det ärftliga materialet som överförs från generation till generation. Dessa DNA-sekvenser inkluderar alla gener (den funktionella och fysiska enheten för ärftlighet som överförs från förälder till avkomma) och transkript (RNA-kopiorna som är det första steget i avkodning av den genetiska informationen) som ingår i genomet. Således hänvisar genomik till sekvensering och analys av alla dessa genomiska enheter, inklusive gener och transkript, i en organism. Proteomics, å andra sidan, hänvisar till analysen av den kompletta uppsättningen proteiner eller proteom. Förutom genomik och proteomik finns det många fler områden inom biologi där bioinformatik tillämpas (i.e., metabolomics, transcriptomics). Var och en av dessa viktiga områden inom bioinformatik syftar till att förstå komplexa biologiska system.
många forskare hänvisar idag till nästa våg i bioinformatik som systembiologi, ett tillvägagångssätt för att ta itu med nya och komplexa biologiska frågor. Systembiologi innebär integration av genomik, proteomik och bioinformatikinformation för att skapa en hel systemvy av en biologisk enhet.
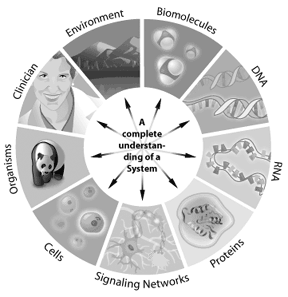
till exempel, hur en signalväg fungerar i en cell kan hanteras genom systembiologi. Generna som är involverade i vägen, hur de interagerar och hur modifieringar förändrar resultaten nedströms, kan alla modelleras med hjälp av systembiologi. Alla system där informationen kan representeras digitalt erbjuder en potentiell applikation för bioinformatik. Således kan bioinformatik tillämpas från enskilda celler till hela ekosystem. Genom att förstå de fullständiga ”dellistorna” i ett Genom får forskare en bättre förståelse för komplexa biologiska system. Att förstå de interaktioner som uppstår mellan alla dessa delar i ett genom eller proteom representerar nästa nivå av komplexitet i systemet. Genom dessa tillvägagångssätt har bioinformatik potential att erbjuda viktiga insikter i vår förståelse och modellering av hur specifika mänskliga sjukdomar eller friska tillstånd manifesterar sig.
början av bioinformatik kan spåras tillbaka till Margaret Dayhoff 1968 och hennes samling av proteinsekvenser som kallas Atlas of Protein Sequence and Structure. Ett av de tidiga signifikanta experimenten inom bioinformatik var tillämpningen av ett sekvenslikhetssökningsprogram för identifiering av ursprunget till en viral gen. I denna studie använde forskare ett av de första sekvenslikhetssökande datorprogrammen (kallad FASTP) för att bestämma att innehållet i v-sis, en cancerframkallande viral sekvens, liknade mest den väl karakteriserade cellulära PDGF-genen. Detta överraskande resultat gav viktiga mekanistiska insikter för biologer som arbetar med hur denna virala sekvens orsakar cancer. Från denna första första tillämpning av datorer till biologi har bioinformatikområdet exploderat. Tillväxten av bioinformatik är parallell med utvecklingen av DNA-sekvenseringsteknik. På samma sätt som utvecklingen av mikroskopet i slutet av 1600-talet revolutionerade biologiska vetenskaper genom att låta Anton Van Leeuwenhoek titta på celler för första gången, har DNA-sekvenseringsteknik revolutionerat bioinformatikområdet. Den snabba tillväxten av bioinformatik kan illustreras av tillväxten av DNA-sekvenser som finns i det offentliga förvaret av nukleotidsekvenser som kallas GenBank.
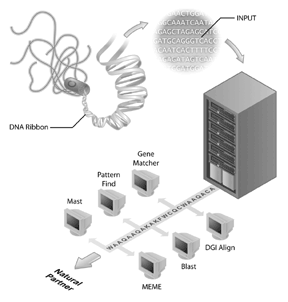
Genomsekvenseringsprojekt har blivit flaggskepp för många bioinformatikinitiativ. Human genome sequencing project är ett exempel på ett framgångsrikt genomsekvenseringsprojekt men många andra genom har också sekvenserats och sekvenseras. Faktum är att de första genomerna som sekvenserades var av virus (dvs., FAG MS2) och bakterier, med genomet av Haemophilus influenzae Rd som det första genomet av en fri levande organism som deponeras i de offentliga sekvensdatabankerna. Denna prestation mottogs med mindre fanfare än slutförandet av det mänskliga genomet men det blir tydligt att sekvenseringen av andra genomer är ett viktigt steg för bioinformatik idag. Emellertid har genomsekvensen i sig begränsad information. För att tolka genomisk information behöver jämförande analys av sekvenser göras och ett viktigt reagens för dessa analyser är de offentligt tillgängliga sekvensdatabaserna. Utan databaser av sekvenser (som GenBank), där biologer har fångat information om deras sekvens av intresse, skulle mycket av den rika informationen som erhållits från genomsekvenseringsprojekt inte vara tillgänglig.
på samma sätt som utvecklingen inom mikroskopi förebådade upptäckter inom cellbiologi, nya upptäckter inom informationsteknologi och molekylärbiologi förebådar upptäckter inom bioinformatik. Faktum är att en viktig del av bioinformatikområdet är utvecklingen av ny teknik som gör det möjligt för vetenskapen om bioinformatik att fortsätta i mycket snabb takt. På datorsidan har Internet, ny mjukvaruutveckling, nya algoritmer och utveckling av datorklusterteknik gjort det möjligt för bioinformatik att göra stora språng när det gäller mängden data som effektivt kan analyseras. På laboratoriumsidan har nya teknologier och metoder liksom DNA-ordnande i viss följd, seriell analys av genuttryck (SAGE), microarrays och nya samlas spectrometrykemistries framkallat på en lika blåsande tempo som möjliggör forskare för att producera data för analyser på en otrolig klassar. Bioinformatik tillhandahåller både plattformsteknologierna som gör det möjligt för forskare att hantera de stora mängder data som produceras genom genomik och proteomikinitiativ samt metoden att tolka dessa data. På många sätt tillhandahåller bioinformatik verktygen för att tillämpa vetenskaplig metod på storskaliga data och bör ses som ett vetenskapligt tillvägagångssätt för att ställa många nya och olika typer av biologiska frågor.
ordet bioinformatik har blivit ett mycket populärt ”buzz” – ord inom vetenskapen. Många forskare tycker att bioinformatik är spännande eftersom det har potential att dyka in i en helt ny värld av okänt territorium. Bioinformatik är en ny vetenskap och ett nytt sätt att tänka som potentiellt kan leda till många relevanta biologiska upptäckter. Även om tekniken möjliggör bioinformatik, handlar bioinformatik fortfarande mycket om biologi. Biologiska frågor driver alla bioinformatiska experiment. Viktiga biologiska frågor kan behandlas av bioinformatik och inkluderar att förstå genotyp-fenotypförbindelsen för mänsklig sjukdom, förstå struktur för att fungera relationer för proteiner och förstå biologiska nätverk. Bioinformatiker finner ofta att de reagenser som är nödvändiga för att svara på dessa intressanta biologiska frågor inte existerar. Således är en stor del av en bioinformatikers jobb att bygga verktyg och tekniker som en del av processen att ställa frågan. För många är bioinformatik mycket populär eftersom forskare kan tillämpa både deras biologi och datorkunskaper för att utveckla reagenser för bioinformatikforskning. Många forskare finner att bioinformatik är ett spännande nytt område för vetenskaplig ifrågasättning med stor potential att gynna människors hälsa och samhälle.
bioinformatikens framtid är integration. Till exempel kommer integration av en mängd olika datakällor som kliniska och genomiska data att göra det möjligt för oss att använda sjukdomssymptom för att förutsäga genetiska mutationer och vice versa. Integrationen av GIS-data, såsom kartor, vädersystem, med grödhälsa och genotypdata, gör det möjligt för oss att förutsäga framgångsrika resultat av jordbruksexperiment. Ett annat framtida forskningsområde inom bioinformatik är storskalig jämförande genomik. Till exempel kommer utvecklingen av verktyg som kan göra 10-vägs jämförelser av genom att driva fram upptäcktsgraden inom detta område av bioinformatik. Längs dessa linjer kan modellering och visualisering av fullständiga nätverk av komplexa system användas i framtiden för att förutsäga hur systemet (eller cellen) reagerar, till exempel på ett läkemedel. En teknisk uppsättning utmaningar står inför bioinformatik och behandlas av snabbare datorer, tekniska framsteg inom disklagringsutrymme och ökad bandbredd, men överlägset en av de största hindren för bioinformatik idag är det lilla antalet forskare inom området. Detta förändras när bioinformatik går i framkant av forskningen men denna fördröjning i expertis har lett till verkliga luckor i kunskapen om bioinformatik i forskarsamhället. Slutligen kommer en viktig forskningsfråga för bioinformatikens framtid att vara hur man beräkningsmässigt jämför komplexa biologiska observationer, såsom genuttrycksmönster och proteinnätverk. Bioinformatik handlar om att omvandla biologiska observationer till en modell som en dator kommer att förstå. Detta är en mycket utmanande uppgift eftersom biologi kan vara mycket komplex. Detta problem med hur man digitaliserar fenotypiska data som beteende, elektrokardiogram och grödhälsa till en datorläsbar form erbjuder spännande utmaningar för framtida bioinformatiker.
(denna artikel är baserad på en intervju med Francis Ouellette, chef för UBC Bioinformatics Center)