La bioinformatique implique l’intégration d’ordinateurs, d’outils logiciels et de bases de données dans le but de répondre aux questions biologiques. Les approches bioinformatiques sont souvent utilisées pour des initiatives majeures qui génèrent de grands ensembles de données. Deux activités importantes à grande échelle qui utilisent la bioinformatique sont la génomique et la protéomique. La génomique fait référence à l’analyse des génomes. Un génome peut être considéré comme l’ensemble complet de séquences d’ADN qui code pour le matériel héréditaire transmis de génération en génération. Ces séquences d’ADN comprennent tous les gènes (l’unité fonctionnelle et physique de l’hérédité transmise du parent à la progéniture) et les transcrits (les copies d’ARN qui constituent l’étape initiale du décodage de l’information génétique) inclus dans le génome. Ainsi, la génomique désigne le séquençage et l’analyse de toutes ces entités génomiques, y compris les gènes et les transcriptions, dans un organisme. La protéomique, d’autre part, fait référence à l’analyse de l’ensemble complet de protéines ou de protéome. En plus de la génomique et de la protéomique, il existe de nombreux autres domaines de la biologie où la bioinformatique est appliquée (i.e., métabolomique, transcriptomique). Chacun de ces domaines importants en bioinformatique vise à comprendre les systèmes biologiques complexes.
De nombreux scientifiques se réfèrent aujourd’hui à la prochaine vague de la bioinformatique sous le nom de biologie des systèmes, une approche pour aborder des questions biologiques nouvelles et complexes. La biologie des systèmes implique l’intégration d’informations génomiques, protéomiques et bioinformatiques pour créer une vue système complète d’une entité biologique.
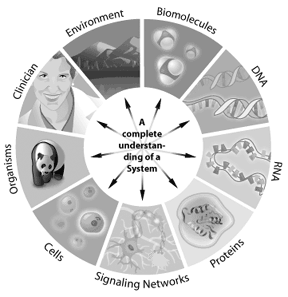
Par exemple, le fonctionnement d’une voie de signalisation dans une cellule peut être abordé par la biologie des systèmes. Les gènes impliqués dans la voie, la façon dont ils interagissent et la façon dont les modifications modifient les résultats en aval peuvent tous être modélisés à l’aide de la biologie des systèmes. Tout système où l’information peut être représentée numériquement offre une application potentielle pour la bioinformatique. Ainsi, la bioinformatique peut être appliquée à partir de cellules individuelles à des écosystèmes entiers. En comprenant les « listes de pièces » complètes d’un génome, les scientifiques acquièrent une meilleure compréhension des systèmes biologiques complexes. Comprendre les interactions qui se produisent entre toutes ces parties d’un génome ou d’un protéome représente le prochain niveau de complexité du système. Grâce à ces approches, la bioinformatique a le potentiel d’offrir des informations clés sur notre compréhension et notre modélisation de la façon dont des maladies humaines spécifiques ou des états sains se manifestent.
Le début de la bioinformatique remonte à Margaret Dayhoff en 1968 et à sa collection de séquences protéiques connue sous le nom d’Atlas de la séquence et de la structure des protéines. L’une des premières expériences significatives en bioinformatique a été l’application d’un programme de recherche de similarité de séquence à l’identification des origines d’un gène viral. Dans cette étude, les scientifiques ont utilisé l’un des premiers programmes informatiques de recherche de similarité de séquence (appelé FASTP), pour déterminer que le contenu de v-sis, une séquence virale cancérigène, était le plus similaire au gène cellulaire PDGF bien caractérisé. Ce résultat surprenant a fourni des informations mécanistiques importantes aux biologistes qui travaillent sur la façon dont cette séquence virale provoque le cancer. Dès cette première application initiale des ordinateurs à la biologie, le domaine de la bioinformatique a explosé. La croissance de la bioinformatique est parallèle au développement de la technologie de séquençage de l’ADN. De la même manière que le développement du microscope à la fin des années 1600 a révolutionné les sciences biologiques en permettant à Anton Van Leeuwenhoek d’examiner les cellules pour la première fois, la technologie de séquençage de l’ADN a révolutionné le domaine de la bioinformatique. La croissance rapide de la bioinformatique peut être illustrée par la croissance de séquences d’ADN contenues dans le référentiel public de séquences nucléotidiques appelé GenBank.
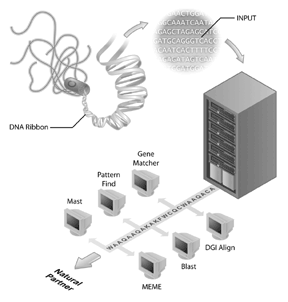
Les projets de séquençage du génome sont devenus les fleurons de nombreuses initiatives en bioinformatique. Le projet de séquençage du génome humain est un exemple de projet réussi de séquençage du génome, mais de nombreux autres génomes ont également été séquencés et sont en cours de séquençage. En fait, les premiers génomes à être séquencés étaient des virus (i.e., le phage MS2) et des bactéries, le génome d’Haemophilus influenzae Rd étant le premier génome d’un organisme vivant libre à être déposé dans les banques de données de séquences publiques. Cette réalisation a été reçue avec moins de succès que l’achèvement du génome humain, mais il devient clair que le séquençage d’autres génomes est une étape importante pour la bioinformatique aujourd’hui. Cependant, la séquence du génome en elle-même a des informations limitées. Pour interpréter l’information génomique, une analyse comparative des séquences doit être effectuée et un réactif important pour ces analyses sont les bases de données de séquences accessibles au public. Sans les bases de données de séquences (telles que GenBank), dans lesquelles les biologistes ont capturé des informations sur leur séquence d’intérêt, une grande partie des informations riches obtenues grâce aux projets de séquençage du génome ne seraient pas disponibles.
De la même manière que les développements en microscopie préfiguraient les découvertes en biologie cellulaire, les nouvelles découvertes en technologies de l’information et en biologie moléculaire préfigurent les découvertes en bioinformatique. En fait, une partie importante du domaine de la bioinformatique est le développement de nouvelles technologies qui permettent à la science de la bioinformatique de se dérouler à un rythme très rapide. Côté informatique, Internet, les nouveaux développements logiciels, les nouveaux algorithmes et le développement de la technologie des grappes informatiques ont permis à la bioinformatique de faire de grands pas en termes de quantité de données pouvant être analysées efficacement. Du côté des laboratoires, de nouvelles technologies et méthodes telles que le séquençage de l’ADN, l’analyse en série de l’expression génique (SAGE), les puces à puces et les nouvelles chimies de spectrométrie de masse se sont développées à un rythme tout aussi fulgurant, permettant aux scientifiques de produire des données pour des analyses à un rythme incroyable. La bioinformatique fournit à la fois les technologies de plate-forme qui permettent aux scientifiques de traiter les grandes quantités de données produites par les initiatives de génomique et de protéomique, ainsi que l’approche pour interpréter ces données. À bien des égards, la bioinformatique fournit les outils nécessaires pour appliquer une méthode scientifique à des données à grande échelle et doit être considérée comme une approche scientifique permettant de poser de nombreux types de questions biologiques nouveaux et différents.
Le mot bioinformatique est devenu un mot « buzz » très populaire en science. De nombreux scientifiques trouvent la bioinformatique passionnante car elle offre le potentiel de plonger dans un tout nouveau monde de territoires inexplorés. La bioinformatique est une nouvelle science et une nouvelle façon de penser qui pourrait potentiellement mener à de nombreuses découvertes biologiques pertinentes. Bien que la technologie permette la bioinformatique, la bioinformatique est toujours une question de biologie. Les questions biologiques animent toutes les expériences de bioinformatique. Les questions biologiques importantes peuvent être abordées par la bioinformatique et comprennent la compréhension de la connexion génotype-phénotype pour la maladie humaine, la compréhension des relations structure-fonction pour les protéines et la compréhension des réseaux biologiques. Les bioinformaticiens constatent souvent que les réactifs nécessaires pour répondre à ces questions biologiques intéressantes n’existent pas. Ainsi, une grande partie du travail d’un bioinformaticien consiste à construire des outils et des technologies dans le cadre du processus de pose de la question. Pour beaucoup, la bioinformatique est très populaire car les scientifiques peuvent appliquer leurs compétences en biologie et en informatique au développement de réactifs pour la recherche en bioinformatique. De nombreux scientifiques constatent que la bioinformatique est un nouveau territoire passionnant de questionnements scientifiques avec un grand potentiel pour la santé humaine et la société.
L’avenir de la bioinformatique est l’intégration. Par exemple, l’intégration d’une grande variété de sources de données telles que des données cliniques et génomiques nous permettra d’utiliser les symptômes de la maladie pour prédire les mutations génétiques et vice versa. L’intégration de données SIG, telles que des cartes, des systèmes météorologiques, avec des données sur la santé des cultures et le génotype, nous permettra de prédire les résultats positifs des expériences agricoles. Un autre futur domaine de recherche en bioinformatique est la génomique comparative à grande échelle. Par exemple, le développement d’outils capables de faire des comparaisons à 10 voies de génomes fera progresser le taux de découverte dans ce domaine de la bioinformatique. Dans ce sens, la modélisation et la visualisation de réseaux complets de systèmes complexes pourraient être utilisées à l’avenir pour prédire comment le système (ou la cellule) réagit, à un médicament par exemple. La bioinformatique est confrontée à un ensemble de défis techniques auxquels sont confrontés des ordinateurs plus rapides, des avancées technologiques en matière d’espace de stockage sur disque et une bande passante accrue, mais l’un des plus grands obstacles auxquels la bioinformatique est aujourd’hui confrontée est le petit nombre de chercheurs dans le domaine. Cela change à mesure que la bioinformatique passe à l’avant-garde de la recherche, mais ce retard dans l’expertise a entraîné de réelles lacunes dans les connaissances en bioinformatique dans la communauté de la recherche. Enfin, une question de recherche clé pour l’avenir de la bioinformatique sera de savoir comment comparer par calcul des observations biologiques complexes, telles que les modèles d’expression des gènes et les réseaux de protéines. La bioinformatique consiste à convertir les observations biologiques en un modèle qu’un ordinateur comprendra. C’est une tâche très difficile car la biologie peut être très complexe. Ce problème de numérisation des données phénotypiques telles que le comportement, les électrocardiogrammes et la santé des cultures sous une forme lisible par ordinateur offre des défis passionnants pour les futurs bioinformaticiens.
(Cet article est basé sur une entrevue avec Francis Ouellette, directeur du Centre de bioinformatique de l’UBC)